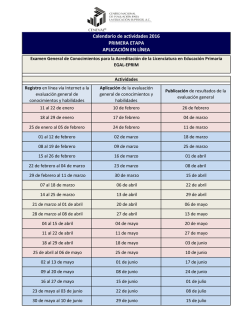
Informes de los participantes
Participantes Lunes 7 de Diciembre del 2015 Grupo de Alberto Darszon (2014-2015) Título: Permeabilidad iónica y comportamiento en el espermatozoide. Resumen: La regulación de la permeabilidad iónica membranal de los gametos es crucial para que estos se fusionen y ocurra la fecundación. Los canales iónicos participan importantemente en la movilidad, maduración e inducción de un proceso exocitótico que prepara al espermatozoide para fusionarse con el óvulo llamado la reacción acrosomal (RA). Además de ser fundamental para la preservación de las especies, la fecundación es importante en la salud humana, la ganadería y la pesca. La movilidad del espermatozoide del erizo de mar está regulada por péptidos pequeños de la capa de gelatina que rodea al óvulo de este animal marino, un modelos preferido para estudiar la fecundación y la quimiotaxis. Registrando simultáneamente la trayectoria, la forma del flagelo y las concentración de Ca2+ intracelular ([Ca2+]i), establecimos que el speract, un decapéptido del óvulo homólogo, induce fluctuaciones en el [Ca2+]i que regulan el nado del espermatozoide. Descubrimos que gradientes de speract disparan incrementos en la [Ca2+]i regulados en tiempo y espacio que desencadenan respuestas quimiotácticas en espermatozoides del erizo de mar de Litechinus pictus. Ahora, en colaboración con el Dr. Guerrero, demostramos por primera vez que gradientes de speract con ciertas características pueden inducir quimiotaxis en espermatozoides de Strongilocentrotus purpuratus. El CatSper, un canal exclusivo del espermatozoide regulado por pHi caracterizado en mamíferos, está en el genoma de S. purpuratus. Resultados experimentales y de modelación recientemente han indicado que CatSper participa en la respuesta al speract. Los primeros registros de pHi con alta resolución temporal en espermatozoides individuales de erizo de mar muestran que el incremento de pHi inducido por el speract inicia en el flagelo y precede a las oscilaciones de Ca2+ que regulan la quimiotaxis. Estos hallazgos indican que el pHi modula las señales de Ca2+ en la respuesta al speract y posiblemente la quimiotaxis, y que, además de CatSper, podría participar otro canal de Ca2+. El espermatozoide de erizo de mar tiene una sola mitocondria, que además de ser la principal fuente de energía, modula los cambios en la [Ca2+]i que determinan su movilidad. Ahora sabemos que el speract induce una depolarización del potencial eléctrico de la membrana interna mitocondrial (ΔΨ) y aumenta los niveles de NADH en espermatozoides de S. purpuratus. Descubrimos que estas respuestas son independientes de Ca2+ y se deben al aumento del pHi inducido por speract que coordina la activación del metabolismo mitocondrial y el nado del espermatozoide para poder fecundar al óvulo homólogo. Actualmente se debate cual es el inductor natural de la RA y donde ocurre esta. Usando una nueva metodología que determina simultáneamente por fluorescencia la RA y los cambios en la [Ca2+]i asociados a esta reacción en el espermatozoide de humano en tiempo real, ahora reportamos nuevos aspectos de esta reacción fundamental en el espermatozoide de ratón. Integrantes del Grupo: Investigadores Asociados Carmen Beltrán Posdoctorales Claudia Sánchez Gerardo Orta Estudiantes de Licenciatura Isela Sarahi Mendoza Rivera Mendoza Estudiantes de Posgrado Enrique Ismael Oliver Santiago-Maestría UAEM Ana Laura Gonzalez Cota-Doctorado M.C. Héctor Ramírez Gómea-Doctorado Juan Garcia Rincón (Dra. Carmen Beltrán) Personal Administrativo Leonel Linares Labastida/Secretario Antonio Blancas/Técnico Miguel A. Trujillo/Técnico de Apoyo Publicaciones: 1. López-González I, Torres-Rodríguez P, Sánchez-Carranza O, Solís-López A, Santi CM, Darszon A., Treviño CL. 2014. Membrane Hyperpolarization during Human Sperm Capacitation. Mol Hum Reprod. [Epub ahead of print] PubMed PMID: 24737063. 2. Darszon A, Hernández-Cruz A. 2014. T-type Ca2+ channels in spermatogenic cells and sperm. Pflugers Arch. 466:819-31. doi: 10.1007/s00424-014-1478-2. PubMed PMID: 24595474. 3. Beltrán C, Rodríguez-Miranda E, Granados-González G, García de De la Torre L, Nishigaki T, Darszon A. 2014. Zn(2+) induces hyperpolarization by activation of a K(+) channel and increases intracellular Ca(2+) and pH in sea urchin spermatozoa. Dev Biol. S00121606(14)00353-4. doi: 10.1016/j.ydbio.2014.07.017. [Epub ahead of print] Review. PubMed PMID: 25092071. 4. Espinal-Enríquez J, Darszon A, Guerrero A, Martínez-Mekler G. 2014. In silico determination of the effect of multi-target drugs on calcium dynamics signaling network underlying sea urchin spermatozoa motility. PLoS One. 9:e104451. doi: 10.1371/journal.pone.0104451. PubMed PMID: 25162222. 5. Silva-Villalobos F, Pimentel JA, Darszon A, Corkidi G. 2014. Imaging of the 3D dynamics of flagellar beating in human sperm. Conf Proc IEEE Eng Med Biol Soc. 2014:190-3. PubMed PMID: 25569929. 6. Sánchez-Cárdenas C, Servín-Vences MR, José O, Treviño CL, Hernández-Cruz A, Darszon A. 2014. Acrosome reaction and Ca ⁺ imaging in single human spermatozoa: new regulatory roles of [Ca ⁺]i. Biol Reprod, 91 (3):67. doi: 10.1095/biolreprod.114.119768. Epub 2014 Aug 6. PubMed PMID: 25100708; PubMed Central PMCID: PMC4435061. 7. Nishigaki T, José O, González-Cota AL, Romero F, Treviño CL, Darszon A. 2014. Intracellular pH in sperm physiology. Biochem Biophys Res Commun. 450(3):1149-58. doi: 10.1016/j.bbrc.2014.05.100. Epub 2014 Jun 2. PubMed PMID: 24887564; PubMed Central PMCID: PMC4146485. 8. Chávez JC, Ferreira JJ, Butler A, De La Vega Beltrán JL, Treviño CL, Darszon A, Salkoff L, Santi CM. 2014. SLO3 K+ channels control calcium entry through CATSPER channels in sperm. J Biol Chem. 2014 Nov 14; 289 (46):32266-75. doi: 10.1074/jbc.M114.607556. PubMed PMID: 25271166; PubMed Central PMCID: PMC4231700. 9. Escoffier J, Navarrete F, Haddad D, Santi CM, Darszon A, Visconti PE. 2015. Flow cytometry analysis reveals that only a subpopulation of mouse sperm undergoes hyperpolarization during capacitation. Biol Reprod. 92(5):121. PMID: 25855261. 10. Vacquier VD, Loza-Huerta A, García-Rincón J, Darszon A, Beltrán C. 2014. Soluble adenylyl cyclase of sea urchin spermatozoa. Biochim Biophys Acta, 1842(12 Pt B):2621-8. doi: 10.1016/j.bbadis.2014.07.011. Epub 2014 Jul 23. PubMed PMID: 25064590; PubMed Central PMCID: PMC4262560. 11. González-Cota AL, Silva PÂ, Carneiro J, Darszon A. 2015. Single cell imaging reveals that the motility regulator speract induces a flagellar alkalinization that precedes and is independent of Ca2+ influx in sea urchin spermatozoa. FEBS Lett, 89(16):2146-54. PMID: 26143372. 12. Navarrete FA, García-Vázquez FA, Alvau A, Escoffier J, Krapf D, Sánchez-Cárdenas C, Salicioni AM, Darszon A, Visconti PE. 2015. Biphasic role of calcium in mouse sperm capacitation signaling pathways. J Cell Physiol. 230(8):1758-69. PMID: 25597298. 13. Ernesto JI, Weigel Muñoz M, Battistone MA, Vasen G, Martínez-López P, Orta G, Figueiras-Fierro D, De la Vega-Beltran JL, Moreno IA, Guidobaldi HA, Giojalas L, Darszon A, Cohen DJ, Cuasnicú PS. 2015. CRISP1 as a novel CatSper regulator that modulates sperm motility and orientation during fertilization. J Cell Biol. 210(7):1213-24. doi: 10.1083/jcb.201412041. PubMed PMID: 26416967; PubMed Central PMCID: PMC4586743. 14. Sánchez-Carranza O, Torres-Rodríguez P, Darszon A, Treviño CL, López-González I. 2015. Pharmacology of hSlo3 channels and their contribution in the capacitation-associated hyperpolarization of human sperm. Biochem Biophys Res Commun. 466(3):554-9. doi: 0.1016/j.bbrc.2015.09.073. Epub 2015 Sep 14. PubMed PMID: 26381170. 15. López-González I, Treviño CL, Darszon A. 2016. Regulation of Spermatogenic Cell T-Type Ca2+ Currents by Zn2+: Implications in Male Reproductive Physiology. J Cell Physiol. 231(3):659-67. doi: 10.1002/jcp.25112. PubMed PMID: 26222306. Donativos: 1. CONACyT de grupo 128566 “Canales de Ca2+ y Cl-, e intercambiadores Cl-/HCO3involucrados en la movilidad, maduración y reacción acrosomal del espermatozoide”. 20102014. 2. DGAPA IN-202312. Canales de Ca2+, K+ y de Cl- que participan en la capacitación del espermatozoide de ratón y humano. 2012-2014. 3. NIH RO1 Ro1 HD038082. Cross talk between Ca2+ and other pathways in capacitation. PI Pablo Visconti/Darszon. 2014-2018. Participación en docencia: Curso de Biología Celular, 2 clases por semestre. Taller de Facultad de Ciencias I, una clase por año. Tópico de Análisis de Imágenes del 2o semestre 2015. Títulos y autores de los carteles que presentaran el grupo: 1. Strongylocentrotus purpuratus sperm accumulate only in steep chemoattractant gradients Ramírez-Gómez H., Jiménez V., Darszon A., Guerrero A. 2. Electrophysiological and pharmacological characterization of Slo3 channel in mouse spermatogenic cells. Verónica Loyo Celis, Alberto Darszon, Gerardo José Orta Salaza. Grupo de Edmundo Calva *Se reporta el período 2013-2015, debido a que no presenté el año pasado por motivo de sabático Título: LeuO y OmpR en el mundo quiescente Resumen: Los esfuerzos de investigación por los miembros de nuestro laboratorio han resultado en el descubrimiento de las porinas OmpS1 y OmpS2, en el regulón LeuO, y en el regulador LtrR en Salmonella Typhi, el agente causal de la fiebre tifoidea. Los genes ompS1, ompS2 y leuO son quiescentes; esto es, se expresan a niveles muy bajos en condiciones de crecimiento estándar de laboratorio (37oC, medio rico, aireación fuerte), y sin embargo se pueden expresar a niveles mucho más altos bajo otras condiciones. Las porinas constituyen antígenos importantes en Salmonella y su función más estudiada es como formadoras de poros en la membrana externa, en donde participan en la exclusión de compuestos tóxicos. En contraste a OmpS1 and OmpS2, las porinas mayoritarias OmpC and OmpF se expresan a niveles muy altos en la célula, siendo de las proteínas más abundantes bajo condiciones estándar de laboratorio. La razón para este contraste no es aparente todavía. LeuO en Typhi regula la expresión de un grupo ecléctico de genes, que incluyen a ompS1, ompS2, assT, y a la agrupación de casA-E-cas1-2-crispr. En el caso de ompS1, nuestro grupo ha mostrado que LeuO antagoniza el efecto represivo por la proteína nucleoide HNS. La acción de LeuO permite que OmpR-P regule positivamente la expresión de ompS1 y ompS2; de otra manera OmpR-P no tiene un efecto, a pesar de ser un regulador global. Otros genes que implican la acción concertada de LeuO y de otros reguladores son materia de investigaciones actuales. De cualquier manera, una de las principales preguntas en el campo se refiere a cuáles son las señales ambientales y reguladores que conllevan a la expresión del gen leuO. Realizamos una caracterización bioquímica y genética de la proteína LeuO, habiendo encontrado que forma tetrámeros activos y que cada uno de sus dominios parece actuar de manera sinérgica para mantener sus funciones reguladoras. Hemos contribuido -junto con el grupo dirigido por Constantino López-Macías- hacia la elucidación de OmpS1 and OmpS2 como inmunógenos potentes y protectores con propiedades adyuvantes. Esto, junto con el hecho que las cepas ompS1 y ompS2 de Typhimurium están atenuadas para la virulencia en el ratón, refuerza el carácter quiescente de estos genes, que deben tener una función en patogénesis. Al regulador LtrR lo denominamos por el inglés “LysR-type OmpR regulator”, y tiene jerarquía sobre el OmpR. De esta manera, nuestro grupo ha descrito la cascada genética formada por ltrR-ompR-ompC, que determina la resistencia al deoxicolato (una sal biliar), y por tanto hemos encontrado un nuevo papel para la porina OmpC. La descripción de LtrR ha abierto la posibilidad de muchos otros estudios sobre la función y regulación de OmpR. Más aún, ltrR y ompR se expresan a niveles mucho más altos cuando la bacteria crece en medio mínimo, en contraste al medio rico, abriendo formalmente la posibilidad de que ompR y muchos otros genes son ciertamente quiescentes, dependiendo de las señales ambientales. Finalmente, nuestro grupo ha publicado las primeras secuencias completas de varios genomas de cepas mexicanas de Salmonella Typhimurium, que son multirresistentes a antibióticos y de relevancia clínica. Esto es, hemos ido del paciente al genoma del patógeno. Estas cepas contienen muchos plásmidos y profagos, y al menos una de ellas una gran cantidad de pseudogenes. Sin embargo, no contienen el plásmido de virulencia y no son patógenas para el ratón, aunque lo son para el humano, siendo incluso algunas invasivas. Esto abre nuevas preguntas sobre cómo el genoma accesorio (plásmidos y profagos) participa en la patogénesis, posiblemente a través de genes quiescentes. Integrantes del grupo Personal Académico 1. Dr. Ismael Hernández Lucas, Inv. Tit. B 2. Dr. Víctor Bustamante Santillán, Inv. Tit. B (desde nov 2014) 3. Dr. Ricardo Oropeza Navarro, Inv. Tit. A (hasta oct 2014)* 4. Dra. Claudia Verónica Silva Romero, Inv. Postdoctoral (ECM) 5. Dr. José Miguel Villarreal Ascencio, Inv. Postdoctoral (IHL)* 6. Dra. Deyanira Pérez Morales, Inv. Postdoctoral (VHB)* 7. M. en C. Marcos Fernández Mora, Téc. Acad. Tit. C 8. Dra. Alejandra Vázquez Ramos, Téc. Acad. Tit C *Terminaron su estancia en este periodo Estudiantes 1. Carmen Guadarrama Román, doctorado (ECM). 2. Liliana Medina Aparicio, doctorado (IHL). 3. Javier Esteban Rebollar Flores, doctorado (IHL). 4. María Magdalena Banda Hernández, doctorado (VHB). 5. Claudia Cristina Paredes Amaya, doctorado (VHB). 6. Sergio Jahir Flores Lozano, maestría (ECM). 7. Diego Sánchez Popoca, maestría (ECM). 8. Lorena Zavala García, maestría (IHL). 9. Noé Becerra Lobato, maestría (IHL). 10. José Crispín Zavala Alvarado, maestría (VHB). 11. Víctor Osio, licenciatura (IHL). 12. América Abigail Beltrán Luviano, licenciatura (IHL). Personal Administrativo 1. Lic. Amapola Blanco Zabala. Gerente de Proyectos 2. Srita. Rosalva González Arenas, Secretaria Publicaciones 1. Moreno-Eutimio, M., Tenorio-Calvo, A., Pastelín-Palacios, R., Pérez-Shibayama, C., Gil-Cruz, C., López-Santiago, R., Baeza, I., Fernández Mora, M., Bonifaz, L., Isibasi, A., Calva, E., and López-Macías, C. 2013. “Salmonella Typhi OmpS1 and OmpS2 porins are potent protective immunogens with adjuvant properties.” Immunology 139: 459-471. 2. Wiesner, M., Fernández-Mora, M., Cevallos, M.A., Zavala-Alvarado, C., Zaidi, M.B., Calva, E., and Silva, C. 2013. “Conjugative transfer of an IncA/C plasmid-borne blaCMY2 gene through genetic re-arrangements with an IncX1 plasmid.” BMC Microbiology 13: 264. 3. Silva, C., Calva E., and Maloy, S. 2014. “One Health and foodborne disease: Salmonella transmission between humans, animals, and plants.” Microbiology Spectrum 2(1): OH-0020-2013. doi:10.1128/microbiolspec.OH-0020-2013 4. Bustamante, V. H., and Calva, E. 2014. “LeuO, a dormant sentinel for SPI-1?” Molecular Microbiology. 91: 1054-1056. 5. Flores-Valdez, M.A., Fernández-Mora, M., Ares, M. Á, Girón, J.A., Calva, E., and De la Cruz, M.Á. 2014. “OmpR phosphorylation regulates ompS1 expression by differentially controlling the use of promoters.”Microbiology-SGM, 160: 733-741. 6. Guadarrama, C., Medrano-López, A., Oropeza, R., Hernández-Lucas, I., and Calva, E. 2014. “The Salmonella Typhi LeuO global regulator forms tetramers: residues involved in oligomerization, DNA binding and transcriptional regulation.” Journal of Bacteriology 196: 2143-2154. 7. Villarreal, J. M., Becerra-Lobato, N., Rebollar-Flores, J.E., Medina-Aparicio, L., Carbajal-Gómez, E., Zavala-García, M.L., Vázquez, A., Gutiérrez-Ríos, R.M., Olvera, L., S. Encarnación, S., Martínez-Batallar, A.G., Calva, E., and Hernández-Lucas, I. 2014. “The Salmonella enterica serovar Typhi ltrR-ompR-ompC-ompF genes are involved in resistance to the bile salt sodium deoxycholate and in bacterial transformation.” Molecular Microbiology 92: 1005-1024. 8. Alvarado-Rodríguez, A., Montañez-Hernández, L. E., Palacio-Molina, S. L., OropezaNavarro, R., Luévanos-Escareño, M.P., Balagurusamy, N. 2014. “Microbial trophic interactions and mcrA gene expression in monitoring of anaerobic digesters.” Frontiers in Microbiology. 5: 597. 9. Montañez-Hernández, L. E., Oropeza Navarro, R., Sierra-Campos, E., De la FuenteSalcido, N. M., y Balagurusamy, N. 2014. “Metanógenos: Una perspectiva de su fisiología con respecto a evolución de la vida”. En: Fronteras en Microbiología Aplicada. Universidad Autónoma de Coahuila. 151-184. ISBN: 978-607-506-204-4. 10. Ipinza, F., Collao, B., Monsalva, D., Bustamante, V.H., Luraschi, R., Alegría-Arcos, M., Almonacid, D.E., Aguayo, D., Calderón, I.L., Gil, F., Santiviago, C.A., Morales, E.H., Calva, E., and Saavedra, C.P. 2014. “Participation of the Salmonella OmpD porin in the infection of RAW264.7 macrophages and BALB/c mice.” PLoS One 9: (10) e111062. /doi: 10.1371/journal.pone.0111062. 11. Guadarrama, C., Villaseñor, T., and Calva, E. 2014. “The subtleties and contrasts of the LeuO regulator in Salmonella Typhi: the implications in the immune response.” Frontiers in Immunology 5: 581. /doi: 10.3389/fimmu.2014.00581 12. Cubillas, C. Vinuesa, P. Tabche, M. L. Dávalos, A. Vázquez, A. Hernández-Lucas, I. Romero, D. García-de-los-Santos, A. 2014. The cation diffusion facilitator protein EmfA of Rhizobium etli belongs to a novel subfamily of Mn/Fe transporters conserved in alphaproteobacteria. Metallomics, 6, 1808-1815. 13. Martínez, L.C., Banda, M.M., Fernández-Mora, M., Santana, F.J., and Bustamante, V.H. 2014. HilD induces expression of Salmonella Pathogenicity Island 2 genes by displacing the global negative regulator H-NS from ssrAB. J. Bacteriol. 196:3746-3755. 14. Martínez, L.C., Martínez-Flores, I., Salgado, H., Fernández-Mora, M., Medina-Rivera, A., Puente, J.L., Collado-Vides, J., and Bustamante, V.H. 2014. In silico identification and experimental characterization of regulatory elements controlling the expression of the Salmonella csrB and csrC genes. J. Bacteriol. 196:325-336. 15. Oropeza, R., Salgado-Bravo, R., and Calva, E. 2015. “Deletion analysis of RcsC reveals a novel signaling pathway controlling polyglucosamine synthesis and biofilm formation in Escherichia coli.” Microbiology-SGM 161: 903-913. 16. Calva, E., Silva, C., Zaidi, M., Sánchez-Flores, A., Estrada, K., Silva, G., SotoJiménez, M., Wiesner, M., Fernández-Mora, M., Edwards, R., and Vinuesa, P. 2015. “Complete genome sequencing of a multidrug-resistant and human-invasive Salmonella enterica serovar Typhimurium strain of the emerging ST213 genotype.” Genome Announcements May/June 2015; 3:e00663-15; doi:10.1128/genomeA.00663-15 17. De la Cruz, M.A., Pérez-Morales, D., Palacios, I.J., Fernández-Mora, M., Calva E., and Bustamante, V.H. 2015. “The two-component system CpxR/A represses the expression of Salmonella virulence genes by affecting the stability of the transcriptional regulator HilD.” Front. Microbiol. 6:807. doi: 10.3389/fmicb.2015.00807 18. Gutiérrez-Huante, M., Martínez, H., Bustamante, V.H., Puente, J.L., and Sánchez, J. 2015. Bicarbonate enhances the in vitro antibiotic activity of kanamycin in Escherichia coli. Lett. Appl. Microbiol. 60:440-446. 19. Martínez-Gamboa, A., Silva, C., Fernández-Mora, M., Wiesner, M., Ponce de León, A., and Calva, E. 2015. “IS200 and multilocus sequence typing for the identification of Salmonella enterica serovar Typhi strains from Indonesia.” International Microbiology 18: 99-104. 20. Silva, C., Calva, E., Calva, J.J., Wiesner, M., Fernández-Mora, M., Puente, J.L., and Vinuesa. 2015. “Complete genome sequence of a human-invasive Salmonella enterica serovar Typhimurium strain of the emerging sequence type 213 harboring a multidrug resistance IncA/C plasmid and a blaCMY-2-carrying IncF plasmid.” Genome Announcements November/December 2015 3:e01323-15; doi:10.1128/genomeA.01323-15 21. Hernández, V. M. Girard, L. Hernández-Lucas, I. Vázquez, A. Ortiz-Ortiz, C. Díaz, R. Dunn, M. F. 2015. Genetic and biochemical characterization of arginine biosynthesis in Sinorhizobium meliloti 1021. Microbiology, 161, 1671-1682. 22. Ramírez-Trujillo, J. A. Dunn, M.F. Suarez-Rodríguez, R. Hernández-Lucas, I. 2015. The Sinorhizobium meliloti glyoxylate cycle enzyme isocitrate lyase (AceA) is required for the utilization of poly-β-hydroxybutyrate during carbon starvation. Annals of Microbiology, Jul 24. 2015 eprint first. Corresponding author Ismael Hernández Lucas. 23. “A multi-drug resistant Salmonella Typhimurium ST213 human-invasive strain (33676) containing the blaCMY-2 gene on an IncF plasmid is attenuated for virulence in BALB/c mice” Wiesner, M., Calva, J.J., Bustamante, V.H., Pérez-Morales, D., FernándezMora, M., Calva, E., and Silva, C. BMC Microbiology (accepted). Alumnos graduados 1. Ana Lucía Gallego Hernández, doctorado, 2014 (tutor E. Calva) 2. María Magdalena Banda Hernández, maestría, 2014 (tutor V.H. Bustamante) 3. Rubiceli Manzo Durán, maestría, 2015 (tutor V.H. Bustamante) 4. Nayeli Alvarado Medina, licenciatura, 2014 (tutor M. Fernández Mora) 5. Andrea Teresa Téllez Galicia, licenciatura, 2014 (tutor M. Fernández Mora) 6. Myriam Estefanía Peña Zúñiga, licenciatura, 2014 (tutor R. Oropeza) 7. Diego Sánchez Popoca, licenciatura, 2015 (tutor M. Fernández Mora) Participación en docencia - Tópico selecto del posgrado en Ciencias Bioquímicas “Mecanismos de regulación de la expresión génica en bacterias”, IBT, UNAM. 2014-2 y 2015-2. Responsable, Víctor H. Bustamante junto con la Dra. Cinthia Núñez. - Participación en el curso de Biología Molecular de la Maestría en Ciencias Bioquímicas. Instituto de Biotecnología de la UNAM. Semestres 2014-2, 2015-1, 2015-2, 2016-1. Cinco clases por semestre. Víctor H. Bustamante Donativos 1. Conacyt núm 179946, 2012-2015. E. Calva 2. PAPIIT/DGAPA 201513, 2012-2015. E. Calva 3. PAPIIT/DGAPA IN204513. “Generación y caracterización de bancos de mutantes afectados en formación de biopelículas en Escherichia coli” Enero de 2013 a diciembre de 2015. Ricardo Oropeza. 4. CONACYT/Ciencia Básica 179071. “Integración y dinámica de la red de regulación que controla la expresión de los regulones de virulencia SPI-1 y SPI-2 de Salmonella enterica” enero de 2013 a diciembre de 2015. Víctor H. Bustamante. 5. PAPIIT/DGAPA IN203415. “Regulación de genes ancestrales por el sistema de dos componentes SsrA/B: un mecanismo de evolución de la patogenicidad de Salmonella enterica” Enero de 2015 a diciembre de 2017. Víctor H. Bustamante. Títulos y autores de los carteles que presentará el grupo: 1) "Descifrando funcionalmente el genoma de Salmonella Typhi", por Ismael Hernández Lucas, José Miguel Villarreal Ascencio, Liliana Medina Aparicio, Javier Esteban Rebollar Flores, Lorena Zavala García, Noé Becerra Lobato, Víctor Osio, América Abigail Beltrán Luviano, Edmundo Calva. 2) "Mecanismo de regulación que funciona como un interruptor de virulencia en Salmonella", por Víctor H. Bustamante, Deyanira Pérez, María M. Banda, Heladia Salgado, Irma Martínez, José A. Ibarra, Edmundo Calva. 3) "Diversidad y evolución de Salmonella Typhimurium de México: el papel del genoma accesorio", por Claudia Silva, Magdalena Wiesner, Marcos Fernández-Mora, Juan J. Calva, Miguel A. Cevallos, Rob Edwards, Víctor H. Bustamante, Alejandro SánchezFlores, José L. Puente, Pablo Vinuesa, Edmundo Calva. Grupo de Alejandra A. Covarrubias (2014-2015) Título: Mecanismos moleculares de la respuesta al déficit hídrico en plantas. Resumen: El interés de nuestro grupo ha sido identificar y tratar de entender los mecanismos de respuesta de plantas vasculares a condiciones de limitación de agua. En estos dos últimos años hemos avanzado principalmente en los proyectos dirigidos a: (a) identificar y determinar la función de microRNAs en la respuesta a la limitación de agua en frijol. En este período se obtuvieron avances relacionados con la caracterización funcional de algunos de los microRNAs identificados como específicos de leguminosas. El análisis informático de una biblioteca del ‘degradoma’ permitió identificar o corroborar los blancos de los microRNAs de interés, Así mismo permitió identificar nuevos microRNAs o nuevos blancos para microRNAs ya descritos. En estos dos últimos años se ha avanzado en caracterización de la función de los miRNAs y sus RNAs blanco en la respuesta del frijol al déficit hídrico, principalmente utilizando el sistema de transformación transitoria en raíces pilosas Así, se han determinado los procesos de biogénesis de los microRNAs de interés lo que ha demostrado la participación de otros RNAs pequeños como responsables de su papel regulatorio. También se han identificado mecanismos novedosos de procesamiento para algunos precursores que implican un procesamiento alternativo, en particular para aquél que codifica para dos microRNAs cuyos transcritos blancos son diferentes. La participación de otro microRNA en el control del crecimiento de la hoja de frijol bajo condiciones de estrés también ha resultado de interés. Se iniciraon proyectos relacionados con la caracterización de microRNAs en otra leguminosa modelo como lo es Medicago truncatula y en una planta perteneciente a las Briofitas, específicamente, Hepáticas llamada Marchantia polymorpha, la cual es un representante de las primeras especies vegetales que poblaron la tierra tras su evolución en el agua. (b) determinar la participación y la función de las hidrofilinas vegetales durante la respuesta a déficit hídrico. El análisis estructural de diferentes proteínas LEA demostró que estas proteínas poseen un alto grado de desorden estructural en solución acuosa y que son capaces de adoptar conformaciones ordenadas bajo condiciones de déficit hídrico y/o de amontonamiento molecular. La comparación entre proteínas LEA de diferentes familias reveló que, aunque las proteínas comparadas son desordenadas, su propensión a formar estructuras ordenadas ante baja disponibilidad de agua es diferencial. Hemos avanzado de forma significativa en dilucidar la función y su relación con la estructura en las proteínas LEA del grupo 4; así como en su localización tisular e intracelular. Así como en determinar su interacción con blancos proteícos in vivo, lo cual ha reforzado el mecanismo de acción que hemos propuesto para su funcionamiento. (c) La selección de la regulación de la expresión genética por mecanismos epigenéticos se ha visto favorecida en las plantas como parte de un arreglo complejo de respuestas que implican diferentes niveles de control ante cambios ambientales, debido posiblemente a su estilo de vida sedentario que les ha permitido aclimatarse y adaptarse de forma eficaz. La participación de los RNAs interferentes pequeños hetero-cromáticos (hcsiRNAs) en la modulación del estado epigenético del genom es un tema de interés dado el estilo de vida de las plantas. Sin embargo, poco se conoce sobre la participación de hcsiRNAs en el control de la respuesta al déficit hídrico de las plantas. Nosotros nos hemos enfocados en conocer la participación de la vía de la metilación de DNA mediada por RNA (RdDM) en esta respuesta al estrés a través de un abordaje genético, lo cual mostró que la deficiencia de diferentes proteínas que participan en esta vía conduce a una respuesta característica de las plantas ante la limitación de agua. Los datos fenotípicos, genéticos y moleculares obtenidos hasta ahora indican que la vía RdDM participan en la modulación de la respuesta a esta condición de estrés y de manera sorpresiva e interesante estos datos sugieren que una de las proteínas centrales de esta vía, AGO4, realiza una actividad adicional a la que s ele ha asignado en esta vía de regulación. (d) caracterizar factores transcripcionales implicados en la respuesta a déficit hídrico en frijol. En este caso se logró identificar todos los factores transcripcionales tipo DREB codificados en el genoma de esta especie, lo cuales participan en la vía de regulación de la respuesta a esta condición de estrés de manera independiente a la vía de transducción mediada por ácido abscísico (ABA). Se obtuvieron además los patrones de acumulación de transcritos por qRTPCR bajo condiciones óptimas de crecimiento y bajo diferentes condiciones de estrés. También se generaron plantas transgénicas de Arabidopsis que sobre-producen algunas de estas proteínas para el análisis de sus fenotipos en respuesta a déficit hídrico. La función del subgrupo de los factores de transcripción que actúan como represores está en vía de caracterización. Integrantes del Grupo: Investigadores Alejandra A. Covarrubias Robles (Investigador Titular C) José Luis Reyes Taboada (Investigador Titular B) Francisco Campos Álvarez (Investigador Titular B) Investigadores Pos-doctorales Damaris Godínez Vidal (DGAPA-UNAM) Técnico Académico Rosa María Solórzano Menier Estudiantes Lucero Rivera Nájera (Doctorado CBQ) (Tutor: AAC) César L. Cuevas Velázquez (Doctorado CBQ) (Tutor: AAC) V. Miguel Palomar Olguín (Doctorado CBM) (Tutor: AAC) Guadalupe Sosa Valencia (Doctorado CBM) (Tutor: JLR) Carlos de la Rosa Ureña (Doctorado CBQ) (Tutor: JLR) Beatriz Pérez Morales (Doctorado CBM) (Tutor: JLR) Inti Arroyo Mosso (Maestría CBQ) (Tutor: AAC) David Rendón Luna (Maestría CBQ) (Tutor: AAC) Paulette Romero Pérez (Maestría CBQ) (Tutor: JLR) Coral Martínez Martínez (Maestría CBQ) (Tutor: AAC) Alfonso Sierra Sarabia (Maestría CBQ) (Tutor: JLR) Alma Jenny García Mejía (Licenciatura FC-UNAM; Maestría CBQ) (Tutor: JLR) Esmeralda Analleli Ramirez Velazquez (Licenciatura FES-Iztacalla) (Tutor: FCA) David Garcias Morales (Licenciatura-UJAT) (Tutor: JLR) André Jauma Purcell Ontrup (Licenciatura FC-UNAM) (Tutor: AAC) Título de la presentación: De las proteínas, los microRNAs y la epigenética en las plantas bajo limitación de agua. Publicaciones: 1. Rocío Rodríguez-Valentín, Francisco Campos, Marina Battaglia, Rosa M. Solórzano, Miguel A. Rosales, Alejandra A. Covarrubias. 2014. Group 6 Late Embryogenesis Abundant (LEA) proteins in monocotyledoneos plants: genomic organization and transcript accumulation patterns in response to stress in Oryza sativa. Plant Molecular Biology Reporter. 32:198-208. DOI 10.1007/s11105-013-0641-9. 2. César L. Cuevas-Velazquez, David F. Rendón-Luna, Alejandra A. Covarrubias. 2014. Dissecting the cryoprotection mechanisms for dehydrins. Frontiers in Plant Science 5: 1-6 (artículo No. 583). doi:10.3389/fpls.2014.00583 3. Lucero Y. Rivera-Najera, Gloria Saab-Rincón, Marina Battaglia, Carlos Amero, Nancy O. Pulido, Enrique García-Hernández, Rosa M. Solórzano, José L. Reyes, Alejandra A. Covarrubias. 2014. A group 6 late embryogenesis abundant protein from common bean is a disordered protein with extended helical structure and oligomer-forming properties. Journal of Biological Chemistry. 289: 31995–32009. 4. Sanchez-Díaz I, Rosales-Bravo F, Reyes-Taboada JL, Covarrubias AA, Narvaez-Padilla V, Reynaud E. 2015. The esg gene is involved in nicotine sensitivity in Drosophila melanogaster. PLoS ONE. 10 (7): e0133956. 5. César L. Cuevas-Velazquez, Gloria Saab-Rincón, José L. Reyes, Alejandra A. Covarrubias. The unstructured N-terminal conserved region of Arabidopsis group 4 LEA proteins folds into -helix and is required for protective activity under water deficit. En revisión en Plant Physiology (2015) 6. Formey, D, Iñiguez, LP, Peláez, P, Li, Y-F, Sunkar, R, Sanchez, F, Reyes JL, Hernandez, G 2015. Genome-wide identification of the Phaseolus vulgaris sRNAome using small RNA and degradome sequencing. BMC Genomics, 16: 423. 7. Nova-Franco B, Iñiguez LP, Valdés-López O, Alvarado-Affantranger X, Leija A, Fuentes SI, Ramírez M, Paul S, Reyes JL, Girard L, and Hernández G. 2015. The miR172c-AP2-1 Node as a Key Regulator of the Common Bean - Rhizobia Nitrogen Fixation Symbiosis. Plant Physiol. 168:273-U502. 8. Naya L, Paul S, Valdes-Lopez O, Mendoza-Soto AB, Nova-Franco B, Sosa-Valencia G, Reyes JL, Hernandez G. 2014. Regulation of Copper Homeostasis and Biotic Interactions by MicroRNA 398b in Common Bean. PLoS ONE, 9, e84416. 9. Bueso E., Munoz-Bertomeu J., Campos F., Brunaud V., Martinez L., Sayas E., Ballester P., Yenush L. Serrano R. 2014. Arabidopsis thaliana HOMEOBOX 25 uncovers a role for gibberellins in seed longevity. Plant Physiology. 164, 999-1010. DIVULGACIÓN: AA Covarrubias, JL Reyes, M Battaglia, MA Rosales, S Cuéllar, C Contreras, L Rivera, C De La Rosa, G Sosa, F Rabanal, A Velarde, F Campos, E Ocampo, RM Solórzano. 2014. The response to water deficit in Phaseolus vulgaris. In: Legume Perspectives. The journal of the International Legume Society Legume Society (ISSN2340-1559) 2: 38-41. Entrevistas varias. Alumnos graduados: Licenciatura 1. Coral Martínez Martínez. Estudio sobre la localización tisular e intracelular de la proteína de respuesta a deficit hídrico AtLEA4-5 de Arabidopsis thaliana. Licenciatura en Biología. Facultad de Ciencias. UNAM. 18 de Junio 2014. 2. Esmeralda Analleli Ramirez Velazquez Licenciatura Biología Experimental. FES-Iztacalla. Terminada 2014. Maestría 3. Inti Arroyo Mosso. Estudio de la relación estructura - función de la familia 6 de proteínas LEA. Programa de Maestría en Ciencias Bioquímicas. IBt-UNAM. Febrero 2015. 4. David Rendón Luna. Estudio sobre la redundancia funcional entre algunas proteínas LEA de Arabidopsis thaliana. Programa de Maestría en Ciencias Bioquímicas. IBt-UNAM. 2012. Octubre 2015 5. Paulette Sofía Romero. Análisis de la respuesta a déficit hídrico de los microRNAs de leguminosas en Medicago truncatula. Maestría en Ciencias Bioquímicas, UNAM. Diciembre, 2015. Doctorado 6. Lucero Y. Rivera Nájera. Análisis estructural y funcional de la proteína PvLEA6 de frijol (Phaseolus vulgaris L.). Doctorado en Ciencias Bioquímicas. IBt-UNAM. 2009. 19 marzo, 2015 Participación en docencia (2012-2013): Cursos Licenciatura: Taller “La Biología a partir de las biomoléculas;; nuevos paradigmas y aplicaciones”. 6 h /semestre. Facultad de Ciencias-UNAM. Semestres 14-1, 15-1, 15-2, 16-1. Cursos de Posgrado: José Luis Reyes: 1. Tópico selecto: RNAs pequeños biogénesis y mecanismos de acción. Nivel Posgrado en Ciencias Bioquímicas 3 horas a la semana (Responsable) en Instituto de Biotecnología. Semestres 2015-1, 2016-1. 2. Biología Vegetal, nivel Posgrado en Ciencias Bioquímicas 6 horas al semestre (Responsable, junto con la Dra. H. Porta y F. Sánchez). Instituto de Biotecnología, Semestre 2015-1 y 2016-1. 3. Biología Molecular, nivel Posgrado en Ciencias Bioquímicas 9 horas al semestre (Docente) en Instituto de Biotecnología. Semestres 2015-1 y 2016-1. Francisco Campos A. 1. Biología Celuar. Posgrado en Ciencias. Facultad de Ciencias. UAEM. - Curso de 2. Biología Vegetal. Posgrado en Ciencias Bioquímicas-IBt. UNAM 3. Biología Molecular. Posgrado en Ciencias Bioquímicas-IBt. UNAM Alejandra A. Covarrubias 1. Biología Vegetal (Respuesta de las plantas a estrés abiótico: déficit hídrico, anoxia y calor). Maestría en Ciencias Bioquímicas. 6 horas / semestre. Instituto de Biotecnología. Semestres: 2015-1, 2016-1 2. Tópico de Doctorado. Crecimiento y desarrollo de las hojas de plantas vasculares y el impacto del estrés abiótico. Programa de Doctorado en Ciencias Biomédicas. Semestre 15-2 Donativos: 1. PAPIIT-DGAPA-UNAM (IN-208212).Caracterización del mecanismo de acción de proteínas intrínsecamente desordenadas en la respuesta a la limitación de agua en plantas” Responsable: Alejandra A. Covarrubias. 2012 – 2014. 2. CONACyT - Cooperación Bilateral Científica y Tecnológica, México-Estados Unidos NSF. Structural characterization of proteins involved in the plant response to water déficit, en colaboración con la Dra. Jane Dyson del The Scripps Research Institute (La Jolla, CA EUA). Responsable: Alejandra A. Covarrubias. 2013-2014. 3. PAPIIT- DGAPA-UNAM (IN205112-3). Identificación de transcritos regulados por microRNAs en respuesta a déficit hídrico en Phaseolus vulgaris. 2012-2014. Responsable: José Luis Reyes. 4. CONACyT-SEP. Ciencia Básica CB2010 (151571) Identificación de los transcritos regulados por microRNAs en Phaseolus vulgaris en respuesta a déficit hídrico. 2012-2014. Responsable: José Luis Reyes. 5. CONACyT-SEP. Convocatoria Ciencia Básica 2013. Las hidrofilinas vegetales como un modelo para el estudio de la relación estructura-función de proteínas intrínsecamente desordenadas. Clave: 221448. 2015-2018. Responsable: Alejandra A. Covarrubias. 6. CONACyT-Problemas Nacionales-CONABIO. “La variación genética de las plantas cultivadas en México: estrategias para enfrentar el cambio climático” En consorcio con IBtUNAM, IE-UNAM, IB-UNAM, INEcol, INIFAP, LANGEBIO. 2015-2019. 7. CONACyT-PROGRAMA DE COOPERACION INTERNACIONAL México – Chile Control de la apertura estomática mediada por ácido salicílico en respuesta a infección bacteriana y estrés hídrico en Arabidopsis. Diciembre 2015-Diciembre 2017. Responsable: Alejandra A. Covarrubias. 8. CONACYT. Apoyo al fortalecimiento y desarrollo de Infraestructura 2014. Fortalecimiento del equipo necesario para el estudio de las respuestas de plantas ante distintos estímulos ambientales. Proyecto 225057. Responsible: José Luis Reyes. 9. PAPIIT-DGAPA-UNAM: Regulación de la respuesta a estrés ambiental por microRNAs en plantas terrestres. IN-205015, (2015-2017). Responsable: José Luis Reyes. 10. CONACyT-Fronteras de la Ciencia. Convocatoria 2015-1. La metilación de adenosinas en RNA y su papel en la regulación de la expresión genética en plantas terrestres. Responsable: José Luis Reyes. 11. PAPIIT-DGAPA-UNAM. Identificación de complejos transcripcionales involucrados en la respuesta a estrés por sequía y frío en frijol. IN211914. 2014-2015. Responsable: Francisco Campos. Títulos y autores de los carteles que presentará el grupo: 1. El balance entre orden y desorden en proteínas desordenadas durante la respuesta a déficit hídrico en plantas. César L. Cuevas-Velazquez, Gloria Saab-Rincón, David Rendón-Luna, Coral Martinez C, Inti Arroyo, Dante Cosio, André J. Purcell, José L. Reyes, Alejandra A. Covarrubias. 2. The RdDM pathway is required for an optimal Arabidopsis response to water deficit. Miguel Palomar, Adriana Garay-Arroyo, Alfredo Cruz-Ramírez, Claudia L. Ibarra-Sánchez, Mario Arteaga-Vázquez, José L. Reyes, Alejandra A. Covarrubias 3. A bicistronic microRNA precursor encoding miR398 and miR2119 responds to drought in legumes. Carlos De La Rosa, Alejandra A. Covarrubias, José Luis Reyes Grupo de Enrique Merino (2014-2015) Título: Identificación de señales de regulación mediante genómica comparativa. Resumen: Los seres vivos se caracterizan, entre otras cosas, por su capacidad de responder a estímulos de su exterior. Desde los organismos unicelulares más primitivos, hasta los organismos multicelulares más evolucionados, son capaces de percibir cambios en su medio y reaccionar de una manera apropiada. Muchas de estas respuestas, en un momento determinado, requerirán de cambios en el estado de expresión del genoma: genes que estaban prendidos necesitarán ser apagados y genes latentes necesitarán ser activados. En nuestra plática se presentaran algunos ejemplos teóricos y experimentales para caracterizar elementos de regulación de la expresión genética, tanto en organismos bacterianos como en organismos superiores. Como ejemplo de elementos de regulación de organismos eucariontes, presentaremos nuestro trabajo realizado para la identificación de IRES celulares en levadura. Los IRES son elementos de RNA ubicados en la región 5´UTR que permiten la traducción independiente de CAP (7mGpppN) presentes en aquellos genes que requieren ser traducidos durante ciertas condiciones celulares en la que alguno de los factores de inicio de traducción canónicos han sido modificados, ya sea por cambios en su estado de fosforilación o por proteólisis. Dichas condiciones incluyen la exposición a diferentes tipos de estrés, proliferación celular, angiogénesis, la respuesta a hipoxia e isquemia, apoptosis, oncogénesis y tumorogénesis, entre otros. Presentaremos la estrategia de genómica comparativa y el uso de las Redes Neuronales Artificiales para la identificación de IRES celulares en levaduras y de las aproximaciones estadísticas que permiten evaluar el significado del enriquecimiento de nuestras predicciones en términos de su conservación en grupos definidos de genes ortólogos y en términos de las relaciones existentes en redes de interacciones funcionales entre proteínas de Saccharomyces cerevisiae. En una segunda parte de nuestra plática, mostraremos nuestros primeros resultados dentro de la Biología Sintética consistentes en el diseño de pequeños RNAs reguladores artificiales (arRNAs) y su aplicación en el control en trans de la expresión de diversos genes, incluyendo el de los genes esenciales como el gen rpoD que codifica para el factor sigma 70 en bacterias o el gen ftsZ involucrado en la formación del septo. Mostraremos el diseño de pequeños de RNAs moduladores de la actividad regulatoria de los arRNAs, que permiten que la activación o represión de los arRNAs puede ser revertida. Finalmente, en la última parte de nuestra plática, mostraremos el diseño del primer circuito regulatorio en Biología Sintética con características epigenéticas. Integrantes del Grupo: Investigadores Titulares Dra. Liliana Pardo López. Dra. Rosa Ma. Gutierrez. Dr. Enrique Merino. Técnicos académicos M.B. María Luisa Tabche. Ing. Ricardo Ciria Merce. Estancias Posdoctorales Dra. Diana Sahonero Canavesi Dra. Wendy Itzel Escobedo Hinojosa. Estancia Técnica Dr. Alejandro Estradas Romero. Estudiantes de doctorado Carlos Daniel Vázquez Hernández. Lizeth Soto Ávila. Luis Rodríguez Mejía. Luz Adriana Vega Cabrera. Patricia Oliver Ocano. Zuemy Rodriguez Escamilla. Estudiantes de maestría David Alejandro Abdala Asbun. Karel Estrada Guerra. Paola Alicia Martínez Amador. Estudiante de licenciatura realizando estancias temporales Luis Eduardo Hernández Ortiz. Omar Alejandro Torres Catruita. Servicio Social Vanesa Bahena Villami Publicaciones: 1. Peguero-Sanchez E., Pardo-Lopez L., and Merino E. 2015. IRES-dependent translated genes in fungi: computational prediction, phylogenetic conservation and functional association. BMC Genomics. GICS-D-15-01004R3. Aceptado para su publicación. 2. Doly S., Shirvani H., Gäta G., Meye F.J., Emerit M.B., Enslen H., Achour L., Pardo-Lopez L., Yang S.K., Armand V., Gardette R., Giros B., Gassmann M., Bettler B., Mameli M., Darmon M., Marullo S. 2015. GABAB receptor cell surface export is controlled by an Endoplasmic reticulum gatekeeper. Molecular Psychiatry. doi: 10.1038/mp.2015.72. [Epub ahead of print]. 3. Moran J., Ramirez G., Jimenez L., Cruz A., Perez-Patrigeon S., Hidalgo A., Orozco L., Martinez A., Padilla L., Avila-Moreno F., Cabello C., Granados J., Ortiz-Quintero B., RamirezVenegas A., Ruiz-Palacios G.M., Zlotnik A., Merino E., Zuniga J. 2015. Circulating levels of miR-150 are associated with poorer outcomes of A/H1N1 infection. Experimental and Molecular Pathology, 99, 253-261. 4. Gutierrez-Preciado A., Torres A.G., Merino E., Bonomi H.R., Goldbaum F.A., Garcia-Angulo V.A. 2015. Extensive Identification of Bacterial Riboflavin Transporters and Their Distribution across Bacterial Species. PLoS ONE, 10, e0126124. 5. Camacho M.I., Alvarez A.F., Chavez R.G., Romeo T., Merino E., Georgellis,D. 2015. Effects of the global regulator CsrA on the BarA/UvrY two-component signaling system. Journal of Bacteriology, 197, 983-991. 6. Martinez-Mercado M., Soto-Del Rio M.D., Gutierrez-Rios R.M., Greninger A.L., Contreras J.F., Lopez-Charreton S., Arias C.F., Isa P. 2015. DNA microarray for detection of gastrointestinal viruses. Journal of Clinical Microbiology, 53, 136-145. 7. Grosso-Becerra M.V., Croda-Garcia G., Merino E., Servin-Gonzalez L., Mojica-Espinosa R., Soberon-Chavez G. 2014. Regulation of Pseudomonas aeruginosa virulence factors by two novel RNA thermometers. Proc. Natl. Acad. Sci., 111:15562-15567. 8. Vega-Cabrera, A., Cancino-Rodezno, A., Porta, H., Pardo-Lopez, L. 2014. Aedes aegypti Mos20 Cells Internalizes Cry Toxins by Endocytosis, and Actin Has a Role in the Defense against Cry11Aa Toxin. Toxins. 6, 464-487. 9. Villarreal J.M., Becerra-Lobato N., Rebollar-Flores J.E., Medina-Aparicio L., Carbajal-Gomez E., Zavala-Garcia M.L., Vazquez A., Gutierrez-Rios R.M., Olvera L., Encarnacion S., MartinezBatallar A.G., Calva E., Hernandez-Lucas I. 2014. The Salmonella enterica Serovar Typhi ltrRompR-ompC-ompF Genes are Involved in Resistance to the Bile Salt Sodium Deoxycholate and in Bacterial Transformation Molecular Microbiology, 92, 1005-1024. 10. Cortes-Tolalpa L., Gutierrez-Rios R.M., Martinez L.M., de Anda, R., Gosset G., Bolivar F., Escalante A. 2014. Global transcriptomic analysis of an engineered Escherichia coli strain lacking the phosphoenolpyruvate: carbohydrate phosphotransferase system during shikimic acid production in rich culture médium. Microbial Cell Factories, 13, 28. 11. Paulin L.F., Soto-Del Rio M.D., Sanchez I., Hernandez J., Gutierrez-Rios R.M., LopezMartinez I., Wong-Chew R.M., Parissi-Crivelli A., Isa P., Lopez, S., Arias C.F. 2014. PhyloFlu, a DNA microarray to determine the phylogenetic origin of influenza A gene segments and the genomic fingerprint of viral strains. Journal of Clinical Microbiology, 52, 803-813. 12. Lopez-Leal G., Tabche M.L., Castillo-Ramirez S., Mendoza-Vargas A, Ramirez-Romero M.A., Davila G. 2014. RNA-Seq analysis of the multipartite genome of Rhizobium etli CE3 shows different replicon contributions under heat and saline shock. BMC Genomics, 15, 770. 13. Cubillas C., Vinuesa P., Tabche M.L., Davalos A., Vazquez A., Hernandez-Lucas I., Romero D., Garcia-de-los-Santos A. 2014. The cation diffusion facilitator protein EmfA of Rhizobium etli belongs to a novel subfamily of Mn/Fe transporters conserved in alpha-proteobacteria. Metallomics, 6, 1808-1815. Alumnos Graduados: "Estudio de la dinámica del modelos de competencia en Bacillus subtilis", para nivel Licenciatura. E.Herrera-Delgado Estatus: terminada Carrera o programa académico: Licenciatura en Ciencias Genómicas. Dependencia, Facultad o Escuela: Instituto de Biotecnología. Institución: Universidad Nacional Autónoma de México. Fecha del examen 30/06/2015. Tutor responsable: Rosa María Gutiérrez Ríos. Donativos: 1. CONACYT-SENER-HIDROCARBUROS. 201441 ($37,000,000.00). Implementación de redes de observaciones oceanográficas (físicas, geoquímicas, ecológicas) para la generación de escenarios ante posibles contingencias relacionadas a la exploración y producción de hidrocarburos en aguas profundas del Golfo de México. Septiembre 2014 - Septiembre 2019. Este donativo se adjudicó a un grupo de 6 investigadores del IBT-UNAM del cual la Dra. Liliana Pardo, Dra. Rosa Ma. Gutiérrez y el Dr. Enrique Merino, forman parte. 2. DGAPA-UNAM. IN204515. ($780,000.00). Procesamiento de información y toma de decisiones en el sistema de esporulación de Bacillus subtilis y su corroboración experimental. aprobación, duración 3 años. Clave del Proyecto IN204515. Enero 2015 a Enero 2017. Investigador responsable Dra. Rosa Ma. Gutiérrez. 3. CONACyT-235817. ($1,999,878.00). Desarrollo de un nuevo enfoque computacional para la identificación de sitios de unión de factores transcripcionales bacterianos y su corroboración experimental en miembros de la familia LysR. Abril 2015 –Marzo 2017. Investigador responsable Dr. Enrique Merino. 4. SEP-CONACyT-176381. ($1,200,000). Análisis de la interacción inicial de las toxinas Cry de Bacillus thuringiensis con las células del intestino de Aedes aegypti, su localización durante la infección tardía y la defensa del insecto. Diciembre 2012 a Diciembre 2015. Investigador responsable: Dra. Liliana Pardo López 5. DGAPA-UNAM-IN201012-3. ($600,000). Mecanismo de oligomerización de las toxinas Cry y la participación de proteínas del insecto blanco. Diciembre 2012 a Diciembre 2015. Investigador responsable: Dra. Liliana Pardo López. 6. CONACyT-167585. ($906,222.20). Papel de los factores de transcripción intermodulares en la organización topológicade la red de regulación de Bacillus subtilis. Agosto 2012 a Agosto 2015. Investigador responsable Dra. Rosa Ma. Gutiérrez. 7. DGAPA-UNAM-IN200612-3. ($718,000). Estudio comparativo de la red de regulación trascripcional de Bacillus subtilis, como producto de su análisis en distintas condiciones. Enero 2012 a Enero 2014. Investigador responsable Dra. Rosa Ma. Gutiérrez. 8. CONACyT-154817. ($1,998,358.00). Regulación de la expresión genética basada en riboswitches. Identificación in silico y caracterización funcional, molecular y cinética Enero 2012 - Enero 2014. Investigador responsable Dr. Enrique Merino. 9. SENASICA–SAGARPA. ($1,000,000.00). Generación de Herramientas Bioinformáticas Aplicadas a la Secuenciación de Organismos Genéticamente Modificados. Enero 2013 - Enero 2014. Investigador responsable Dr. Enrique Merino. Participación en docencia: 1. Bases de Programaciónen Perl para Bioinformática. Tópico selecto del Programa de Maestría y Doctorado en Ciencias Bioquímicas. Instituto de Biotecnología-UNAM. 7/Enero/201420/Mayo 2014. Dra. Rosa Ma. Gutiérrez y Dr. Enrique Merino. 2. Bioinformática aplicada para análisis genómicos de microorganismos, secuenciación y anotación genómica, genómica funcional, Posgrado de Doctorado en Ciencias Bioquímicas. UNAM. Agosto/2014 a Diciembre/2014. Dra. Rosa Ma. Gutiérrez y Dr. Enrique Merino. 3. Bases de Programaciónen Perl para Bioinformática. Posgrado de Doctorado en Ciencias Bioquímicas. UNAM. Enero/2015 a Mayo/2015. Dra. Rosa Ma. Gutiérrez y Dr. Enrique Merino. 4. Métodos y estrategias en Bioinformática. Tópico selecto del Programa de Maestría y Doctorado en Ciencias Bioquímicas. UNAM. Agosto/2015 a Diciembre/2015. Dr. Enrique Merino. 5. Ingeniería de vías metabólicas en bacterias. Tópico Selecto Posgrado en Ciencia Bioquímicas 3 horas al semestre (Docente) Impartido en Instituto de Biotecnología de 23/09/2015 a 23/09/2015. Dra. Rosa Ma. Gutiérrez. 6. Temas selectos. Inmunología de las infecciones intracelulares: modulación de respuesta inmune innata y adaptiva por componentes del parásito. Tópico Selecto. Posgrado en Ciencias Biológicas, 3 horas al semestre (Docente) Impartido en Facultad de Medicina de 11/11/2015 a 11/11/2015. Dra. Rosa Ma. Gutiérrez. 7. Tópico de Regulación de la expresión Génica en Bacterias. Maestría en Ciencias UNAM Clases impartidas Traducción (4 horas). 2014, 2015. Dra. Liliana Pardo. 8. Curso básico de Biología Molecular para estudiantes de primer semestre de la maestría en Ciencias Bioquímicas de la UNAM Clases impartidas: Síntesis del Proteoma (3 horas) y Procesamiento y recambio del proteoma (3 horas). 2014, 2015. Dra. Liliana Pardo. Divulgación: 2014. Feria de la Investigación. Puertas abiertas Instituto de Biotecnología UANAM. Dra. Liliana Pardo López, Dra. Rosa Ma. Gutiérrez, Dr. Enrique Merino, M.B. Ma. Luisa Tabche Barrera, Ing. Ricardo Ciria Merce. 2015. Mini-Simposio Avances en la Biotecnología. Conferencia: Los microorganismos que viven en el Golfo de México. Dra. Liliana Pardo López. Lugar: Facultad de Química La Benemérita Universidad Autónoma de Puebla. Febrero 2015. 2015. Conferencia dirigida a estudiantes de Licenciatura: Bacterias que viven el Golfo de México, útiles para descontaminar. Dra. Liliana Pardo López. Lugar: Facultad de Biología, Universidad Veracruzana. Febrero 2015. 2015. Conferencia dirigida a estudiantes de primaria: Virus del Chikungunya. Dra. Liliana Pardo López. Lugar: Escuela Primaria Ocachicualli. Octubre 2015. Títulos y autores de los cárteles que presentará el grupo: 1. Generación de patrones de organización para reacciones y rutas metabólicas. Vázquez Hernández Carlos Daniel y Gutiérrez Ríos Rosa María. 2. Caracterization of Spo0M in Bacillus subtilis: Just a regulator of sporulation? Luz Adriana Vega-Cabrera, María Luisa Tabche, José Luis Rodríguez, Adán Guerrero, Enrique Merino, Liliana Pardo-López Participantes Martes 8 de Diciembre del 2015 Grupo de Enrique Galindo (2014-2015) Título: Ingeniería de Biopolímeros Microbianos Resumen: Desde hace varios años, nuestro grupo de investigación se ha interesado en el estudio de los factores de cultivo que afectan la biosíntesis del polisacárido extracelular alginato y del polímero intracelular polihidroxibutirato (PHB), ambos producidos por Azotobacter vinelandii. Lo anterior con la finalidad de identificar los mecanismos involucrados en la síntesis y la composición química de ambos biopolímeros, con el propósito de mejorar el proceso biotecnológico y eventualmente hacerlo competitivo industrialmente. En este período hemos llevado a cabo estudios sobre el análisis de los principales genes y actividades de las enzimas involucradas en el proceso de polimerización y depolimerización del alginato en condiciones de limitación y no-limitación de oxígeno. Se ha encontrado que en condiciones de oxígeno cercano a cero (limitación), el peso molecular promedio del alginato se incrementa, en comparación a lo que se observa en los cultivos a altos niveles de oxígeno (no-limitación). Este comportamiento está relacionado con un aumento en el nivel de expresión de los genes alg8 y alg44 (involucrados en la polimerización) y una baja actividad de las enzimas depolimerizantes (liasas). Ambos procesos podrían ser los mecanismos mediante los cuales el oxígeno regula la síntesis de alginato de un alto peso molecular (Flores et al., 2015). A partir de esta información hemos establecido condiciones operacionales en cultivos en matraces agitados, para la síntesis de alginatos con un alto peso molecular y alta capacidad viscosificante. También hemos escalado exitosamente el proceso a biorreactores de 3 L, usando como criterio la velocidad de transferencia de oxígeno (Giese et al., 2014) y el consumo de potencia (Gómez-Pazarín et al., 2015). En el caso del PHB, durante el presente período nuestro grupo ha identificado los principales parámetros de fermentación que influyen en los rendimientos y el peso molecular del biopolímero sintetizado por una doble mutante (OPNA) de A. vinelandii (Peña et al. 2014 a y b). Derivado de estos estudios, hemos desarrollado estrategias de cultivo en sistema de cultivo en lote y en lote alimentado, que nos han permitido generar una alta concentración de polímero (35 g/L) con un peso molecular promedio superior a los 3000 kDa (García et al., 2014; Peña et al., 2014 b). Este tipo de polímeros exhiben propiedades termomecánicas y de biocompatibilidad que les confieren ventajas sobre los materiales sintéticos, para ser usados, por ejemplo, como andamios en ingeniería de tejidos (Domínguez-Díaz et al., 2015). En esta misma línea de investigación, más recientemente hemos estudiado la estrecha dependencia que existe entre la velocidad de consumo de oxígeno y la producción de PHB y de alginato, encontrando que las cepas que sólo producen PHB consumen hasta 30 % menos oxígeno que las cepas que sintetizan ambos polímeros (Díaz-Barrera et al., 2015). Desde un punto de vista tecnológico, este tipo de cepas ofrecen ventajas, ya que permitirían alcanzar niveles altos de producción de PHB con un mínimo de aireación en los fermentadores (Peña et al., 2014 b). Integrantes del Grupo: Investigadores Enrique Galindo, Inv. Tit. “C”, SNI III. Leobardo Serrano, Inv. Tit. “B”, SNI II. Carlos Peña, Inv. Tit. “B”, SNI I. Técnico Académico Celia Flores, Técnico Titular “C” Posdoctorado Magdalena Brito (a partir de Septiembre del 2015) Tania Castillo (hasta Julio del 2014) Estudiantes (los graduados en el periodo se indican más adelante) Doctorado Sergio Cristiano (E. Galindo) Andrés García (C. Peña) Modesto Millán (C. Peña) Alehlí Holguín (E. Galindo) Agustín Luna (L. Serrano) Karina Balderas (L. Serrano) Maestría Jonathan Sanguino (C. Peña) Esmeralda Soriano (L. Serrano) Yesenia Malloqui (L. Serrano) Rita García (E. Galindo) Licenciatura Sandra García (E. Galindo) Cynthia Landa (L. Serrano) Rocío Ávila (L. Serrano) Miriam Salazar (C. Peña) Berenice Sandoval (C. Peña) Rafael Sánchez (L. Serrano) Salvador Albavera (C. Peña) Prácticas profesionales/estancias Juan Chávez (C. Flores) Erik Hernández (C. Flores) Roberto Pisté (E. Galindo) Personal de base Leticia Díaz Antonio Dorantes Xochitl González Publicaciones: 1. Bertrand, B., Martínez-Morales, F., Tinoco, J., Rojas, S., Serrano-Carreón, L., TrejoHernández, M.R. 2014. Induction of laccases in Trametes versicolor by aqueous wood extracts”. World Journal of Microbiology and Biotechnology, 30: 135-142. 2. Díaz-Barrera, A., Andler, R., Martínez, I. and Peña, C. 2015. Poly-3-hydroxybutyrate production by Azotobacter vinelandii strains in stirred tanks at different oxygen transfer rates. Journal of Chemical Technology and Biotechnology, DOI: 10.1002/jctb.4548 3. Domínguez-Díaz, M., Romo-Uribe, A., Peña, C., Espín, G., Segura, D., Meneses-Acosta, A. 2015. Thermo-mechanical properties, microstructure and cell viability in poly-βhydroxybutyrates (PHB) produced by OP and OPN strains of Azotobacter vinelandii. European Polymer Journal, 63:101-112. (2015). 4. Flores, C., Díaz-Barrera, A., Martínez, F., Galindo, E., Peña, C. 2015. Role of oxygen in the polymerization and de-polymerization of alginate produced by Azotobacter vinelandii. Minireview, Journal of Chemical Technology and Biotechnology, 90: 356–365. 5. García, A., Segura, D., Espín, G., Galindo, E., Castillo, T. and Peña, C. 2014. High production of poly- -hydroxybutyrate (PHB) by an Azotobacter vinelandii mutant altered in PHB regulation using a fed-batch fermentation process”. Biochemical Engineering Journal, 82: 117123. 6. Giese, H., Klöckner, W., Peña, C., Galindo, E., Lotter., S., Wetzell, K., Meissner, L., Peter, C., Büchs J. 2014. Effective shear rate in shake flasks”. Chemical Engineering Science, 118:102113. 7. Gómez-Pazarín, K., Flores, C., Castillo, T., Büchs, J., Galindo E., Peña C. 2015. Molecular weight and viscosifying power of alginates produced in Azotobacter vinelandii cultures in shake flasks under low power input”. Journal of Chemical Technology and Biotechnology, DOI:10.1002/jctb-4548 (2015). 8. Holguín, A., López, D., Corkidi, G. and Galindo, E. 2015. Foam production and hydrodynamic performance of a traditional Mexican molinillo (beater) in the chocolate beverage preparation process". Food and Bioproducts Processing, 93: 139-147. 9. Peña, C., López, S., García, A., Espín, G., Romo-Uribe, A. Segura, D. 2014a. Biosynthesis of poly-b-hydroxybutyrate (PHB) with a high molecular mass by a mutant strain of Azotobacter vinelandii (OPN)”. Annals of Microbiology, 64: 39-47. 10. Peña, C., Castillo, T., García, A., Segura, D. 2014b. Biotechnological strategies to improve production of microbial polyhydroxybutyrate [P(3HB)]: a review of recent research work. Microbial Biotechnology, 7: 278-293. 11. Rojas-Domínguez, A., Holguín-Salas, A., Galindo, E., Corkidi, G. 2015. Gradient-directionpattern transform for detection of circles -application to automated detection of oil drops in images of multiphase dispersions. Chemical Engineering & Technology, 38(2): 1-10. 12. Tinoco-Valencia, R., Gómez-Cruz, C., Galindo, E., Serrano-Carreón, L. 2014. Toward an understanding of the effects of agitation and aeration on growth and laccases production by Pleurotus ostreatus”. Journal of Biotechnology, 177: 67-73. Capítulos de libro: 1. Romero, Y., Peña, C., Rojas, A., Tortajada, M., Espín, G., Segura, D. 2014. A polyhydroxybutyrate producing mutant of Azotobacter vinelandii lacking the regulatory proteins RsmA and Enzyme IIA NTr utilizes the carbon source more efficiently. En: Bioplásticos, Ed. M Reis and B Méndez. CIYTED, Portugal ISBN: 978-953-307-268-5. Pp. 23-31. 2. Peña, C., García, A., Millán, M., Castillo, T. 2015. Approaches for the synthesis of tailormade polyhydroxyalkanoates (PHAs). En: Microbial Factories: Biodiversity, Biofuels, Biopolymers, Bioactive Molecules and Waste Treatment. Ed. V.C. Kalia, Springer ISBN: 978953-307-268-5. 3. Serrano-Carreón, L., Galindo, E., Rocha-Valadéz, J.A., Holguín-Salas, A. Corkidi, G. “Hydrodynamics, fungal physiology, and morphology”, Advances in Biochemical Engineering and Biotechnology, Ed. T. Bley and R. Krull. Springer International Publishing, Switzerland (2015). 149:55–90 Libro de texto: “A TAREFA DA CIÊNCIA EXPERIMENTAL – Um Guia Prático para Pesquisar e Informar Resultados nas Ciências Naturais”, Enrique Galindo Fentanes, Grupo Editorial Nacional GENLTC. Rio de Janeiro, Brasil. 202 páginas, ISBN 978-85-216-2363-2, 2014. Innovación tecnológica: RESEÑA DE DESARROLLO TECNOLOGICO: “Desarrollo histórico y los retos tecnológicos y legales para comercializar Fungifree AB®, el primer biofungicida 100% mexicano”, E. Galindo, L. Serrano-Carreón, C. R. Gutiérrez, K. A. Balderas-Ruíz, A. L. Muñoz-Celaya, M. Mezo-Villalobos, y J. Arroyo-Colín, TIP Revista Especializada en Ciencias Químico-Biológicas, 18(1):52-60 (2015). AMPLIACIÓN DE LOS REGISTROS DEL BIOFUNGICIDA Fungifree AB para su uso en un total de 20 cultivos diferentes, para el control de antracnosis, cenicilla polvorienta y moho azul y la obtención del registro OMRI para su uso en cultivos orgánicos (2014). E. Galindo y L. Serrano. “Nuevo proceso para la producción de polihidroxibutirato a partir de Azotobacter vinelandii”. Carlos Peña, Daniel Segura, Guadalupe Espín, Tania Castillo y Andrés García. Solicitud de Patente. No de Solicitud en IMPI: MX/a/2014/006115. Alumnos graduados: Doctorado Celia Flores Ocampo (E. Galindo). Maestría Axel Falcón Rojas (E. Galindo). Karen Denisse Gómez Pazarín (C. Peña). Karen Fernández Alejandre (L. Serrano). Alma Lucero Jiménez Patiño (C. Peña). Agustín Luna Bulbanera (L. Serrano). Licenciatura Cristina Gómez Cruz (L. Serrano). Erick Flores Olvera (L. Serrano). Margarita Venegas Brito (L. Serrano). Daniel Gil Cisneros (L. Serrano). Mayra Nieto Ayala (C. Flores). Julio Cesar León Corona (C. Peña). Miseli Pliego Domíguez (C. Peña). Donativos: 1. CONACyT (Problemas Nacionales 247473), E. Galindo, “Integración de desarrollos en biotecnología, automatización y tecnologías de la información para establecer un modelo escalable de producción sustentable e inocua de hortalizas en invernadero” (2015-2017). 2. SEP-CONACyT (CB-2014-01 240438). (Ciencia Básica), E. Galindo, “Entendiendo la inhomogeneidad de la dispersión multifásica y de la transferencia de oxígeno en sistemas modelo y de fermentación, a escala piloto” (2015-2017). 3. PAPIIT-UNAM (IT 201014), E. Galindo, “Análisis local de la dispersión multifásica y área interfacial en un sistema modelo de fermentación micelial de cuatro fases” (2014-2015). 4. CONACyT (IFR 2013-1 204899). L. Serrano, “Actualización y fortalecimiento de equipo nivel planta piloto para el desarrollo de proyectos de investigación, desarrollo tecnológico y formación de recursos humanos en el campo de la ingeniería y escalamiento de bioprocesos” (2014). 5. PAPIIT-UNAM (IN206715). L. Serrano, “Estudio de la contribución de los factores de antagonismo de Bacillus subtilis 83 en el control biológico de Colletotrichum gloeosporioides” (2015-2017). 6. CONACyT (CB-2014-1 240445). L. Serrano, “Establecimiento del papel de la limitación nutricional y de la densidad celular en la producción de lipopéptidos y en la esporulación de Bacillus sp. 83 en cultivos sumergidos” (2015-2017). 7. CONACYT (CB 2014-1-238535). C. Peña, “Producción de polímeros bacterianos por fermentación: estudio del efecto del potencial redox intracelular sobre la biosíntesis de alginato y PHB en cultivos microaerofílicos de Azotobacter vinelandii (2015-2017). 8. CONACyT (CI- 236513). C. Peña, “Desarrollo de un proceso de escalamiento para la producción de bioplásticos bacterianos” (2015-2016). 9. PAPIIT-UNAM (IT 100513). C. Peña, “Estudio del papel del oxígeno disuelto en los procesos de polimerización y depolimerización del polihidroxibutirato (PHB) en cultivos de Azotobacter vinelandii en cultivos en fermentador” (2013-2015). Participación en docencia: 1. Tópico selecto: “Emprendedurismo en el campo de la biotecnología”, Posgrado en Ciencias Bioquímicas, Agosto-Noviembre de 2014 y Agosto-Noviembre de 2015, C. Peña (coordinador), L. Serrano, E. Galindo. 2. Curso “Metodología de la Investigación”, Colegio Marymount (Preparatoria), E. Galindo, Semestre 2014-I (Enero-Junio 2014). 3. Curso-Taller: “Bio-procesos con Microorganismos Recombinantes”, Mayo y Octubre del 2014; Mayo y Octubre de 2015. Organizado por la Planta Piloto del IBt-UNAM. L. Serrano (coordinador), C. Peña, E. Galindo. 4. Curso “Del gen al producto”, Posgrado en Ciencias Bioquímicas, Febrero-Mayo de 2104 y Febrero-Mayo de 2015. Instituto de Biotecnología, UNAM. L. Serrano, C. Peña, E. Galindo. Divulgación: Eventos 1. Biotecnología en Movimiento, Revista de Divulgación del Instituto de Biotecnología en la UNAM, No. 1 (Abril-Junio), No. 2 (Julio-Septiembre), No. 3 (Octubre-Diciembre) 2015. E. Galindo (Editor, con G. Ponce). 2. Primer Día de Puertas Abiertas del IBt (23 de Mayo de 2014), E. Galindo (Coordinador general, con C. Treviño), participación de cerca de 400 personas del IBt, 1,200 visitantes. 3. Día del Ex Alumno del IBt (14 de Noviembre de 2014 y 6 de Noviembre de 2015), E. Galindo (Coordinador general, con M. Trejo y M. Patiño). Publicaciones 1. E. Galindo, G. Corkidi, A. Holguín Salas, D. López López. 2014. Producción de espuma en el chocolate con el molinillo tradicional, Revista Digital Universitaria, 15 (1). 2. El arte de patentar, E. Galindo, sección “Y sin embargo se mueve…”, La Unión de Morelos, 26 de febrero de 2014, pag. 27. 3. La ciencia, desde Morelos para el Mundo, E. Galindo, Sección “Y sin embargo se mueve…”, La Unión de Morelos, 10 de septiembre de 2014, pag. 27. 4. Biopesticide consolidates position in the Mexican market, Serrano-Carreón, L., Galindo, E., Gutiérrez, C.R., Biocontrol News and Information, 35(4), 30N–31N (Diciembre, 2014). 5. Microbios, fermentaciones y biotecnología, Galindo, E. En: F. Rebolledo (Editor y Compilador), Un mundo de bacterias, Instituto de Biotecnología, UNAM Campus Morelos, ISBN: 978-607-02-5179-5, pags. 85-86 (2014). 6. El oficio científico, un ameno y culto libro de Sofía Liberman y Bernardo Wolf, E. Galindo. La Unión de Morelos, 5 de octubre de 2015, pags. 30-31. 7. El Congreso CUAM-ACMor de 2015: veintiseis años promoviendo la investigación hecha por jóvenes, E. Galindo, La Unión de Morelos, 7 de septiembre de 2015, pags. 32-33. 8. Graduación de bachilleres en la comunidad indígena de Cuentepec: logran importante premio de ciencias…y no tienen instalaciones, E. Galindo, Sección “Y sin embargo se mueve…”, La Unión de Morelos, 15 de julio de 2015, pag. 14. 9. Fungifree AB®: el primer biofungicida desarrollado en México, con certificación orgánica y efectivo para el control de enfermedades fungales en 20 cultivos, E. Galindo, L. SerranoCarreón, C.R. Gutiérrez, La Unión de Morelos, 25 de mayo de 2015, pags. 30-31. Conferencias Los integrantes del grupo impartieron cerca de 50 conferencias de divulgación. Distinciones relevantes: 1. PREMIO ADIAT A LA INNOVACIÓN TECNOLÓGICA. 2014, E. Galindo, L. Serrano (con C. R. Gutiérrez), Primer lugar, Categoría PyME, para Agro&Biotecnia S de RL. MI., Asociación Mexicana de Directivos de la Investigación Aplicada y el Desarrollo Tecnológico, A.C. 2. PREMIO “INNOVADORES DE AMERICA 2014, E. Galindo, L. Serrano (con C. R. Gutiérrez) Categoría Empresa e Industria (Agro&Biotecnia), por la innovación “Fungifree AB: el primer biofungicida desarrollado en México”. 3. Huésped Distinguido de la Ciudad de Nuestra Señora de La Paz, 4 noviembre de 2014, La Paz, Bolivia (E. Galindo). Títulos y autores de los carteles que presentará el grupo: 1. Mecanismos difusionales y transcripcionales involucrados en la producción de lacasas por Pleurotus ostreatus CP50. Karen I. Fernández-Alejandre, Noemí Flores, Raunel Tinoco-Valencia, Mario Caro, Celia Flores, Enrique Galindo y Leobardo Serrano-Carreón. 2. Lipopéptidos antifúngicos producidos por Bacillus sp 83. Agustín Luna, Rocío Avila, Gerardo Corzo, Enrique Galindo y Leobardo Serrano-Carreón. Grupo de Francisco Bolivar (2014-2015) Título: Cambios genéticos ocurridos durante el proceso de evolución adaptativa que generó la cepa evolucionada de Escherichia coli PB12. Resumen: La cepa de Escherichia coli JM101 fue inactivada en el sistema de fosfotrasferasa (PTS) dependiente de fosfoenolpiruvato (PEP), con la finalidad de incrementar la disponibilidad intracelular de PEP para redirigirlo a la producción de metabolitos aromáticos de interés industrial, resultando en la cepa denomina PB11 (PTS-) la cual disminuyó de forma importante su capacidad de crecer el glucosa como fuente de carbono respecto a la cepa parental. Esta cepa fue sometida a un proceso de evolución adaptativa en laboratorio (EAL) para incrementar su velocidad específica de crecimiento en glucosa (µ = 0.1 h-1 vs µ = 0.7 h-1, respectivamente). Como resultado de este experimento se aislaron varias cepas evolucionadas que incrementaron su capacidad su capacidad de crecer en glucosa. Dos de estas cepas evolucionadas PB12 y PB13, fueron aisladas a las 120 y 200 horas del experimento de EAL) incrementando su µ en 338 % (0.4 h-1) y 373 % (µ = 0.5 h-1), respectivamente vs su progenitora PB11. La secuenciación y análisis del genoma de ambas cepas evolucionadas y permitió la identificación de una deleción cromosomal de 10,328 pb y dos mutaciones puntuales no sinónimas en los genes dhaM y glpT, los cuales se propone que ocurrieron antes de la divergencia entre las cepas PB12 y PB13, en una etapa temprana del proceso de evolución adaptativa. Entre los genes que se perdieron con la región cromosomal se encuentran, mutH, rppH, aas, lplT y galR, los cuales se propone que están relacionados con el incremento en el fitness en las cepas evolucionadas, causando un incremento en la tasa de mutación de 200 X (mutH), el incremento en la vida media de los RNAm (rppH), el uso de un sistema alternativo de transporte de glucosa (galR) y el incremento en el fitness y mejora de la metabolismo de glucosa al eliminar la estimular la vía de degradación de fosfolípidos por el operón aas‑ lplT. La cepa PB12 ha sido utilizada para la producción de diversos compuestos aromáticos entre los que se encuentran el shikimato. En la actualidad este compuesto se utiliza como la materia prima para la síntesis química del antiviral oseltamivir fosfato, utilizado para el tratamiento de diversos tipos de influenza incluyendo los tipos pandémicos H5N1 y H1N1. La cepa PB12 fue utilizada como fondo genético para la incorporación de modificaciones cromosomales y la construcción de un plásmido que permitió expresar constitutivamente un operón sintético con los genes clave para la síntesis de SHK. La cepa resultante sobreproductora de SHK, AR36 (PB12 lacI- aroKaroL- pykF- trc/aroB+ tktA+ aroGfbr+ aroE+ aroD+ zwf+), exhibió el mayor rendimiento reportado de este compuesto (0.42 mol/mol) y una alta proporción molar respecto a otros subproductos aromáticos (80%). Además, se obtuvo la mayor concentración de SHK alcanzada con un cultivo en lote (43 g/L). Esta cepa puede tolerar y consumir altas concentraciones iniciales de Glc y se encontró una relación entre esta capacidad y la sobreexpresión de genes glucolíticos, de TCA y de asimilación de acetato. Integrantes del Grupo: Académicos Dr. Francisco Bolívar Zapata. Investigador Emérito. Líder Académico (FBZ) Dr. José Adelfo Escalante Lozada. Investigador Titular B (AEL) Dra. Noemí Flores Mejía. Técnico Académico (NFM) MC Ramón de Anda Herrera. Técnico Académico. Estudiantes Doctorado MC José Juan Andrés Martínez Álvarez (FBZ) MC Dulce Catalina Díaz Quiroz (AEL) MC Susy Beatriz Carmona Contreras (AEL) Maestría José de Jesús González Morán (FBZ) Fabián Moreno Avitia (AEL) David Rodrigo López Soto (AEL) Licenciatura David Rodríguez Lozada (FBZ) Naomí Adán Valencia (NFM) Publicaciones: 1. Rodriguez, A., Martinez, J., Flores, N., Escalante, E., Gosset, G., Bolivar, F. 2014. Engineering Escherichia coli to overproduce aromatic amino acids and derived compounds. Microbial Cell Factories. 13:126. DOI:10.1186/s12934-014-0126-z. 2. Cortés-Tolalpa, L., Gutiérrez-Ríos, R. M., Martínez, L. M., de Anda Herrera, R., Gosset, G., Bolívar, F., Escalante, A. 2014 Global transcriptomic analysis of an engineered Escherichia coli strain lacking the phosphoenolpyruvate: carbohydrate phosphotransferase system during shikimic acid production in rich culture medium. Microbial Cell Factories. 13:28 DOI: 10.1186/1475-2859-13-28. ISSN: 1475-2859. 3. Díaz Quiroz, D. C., Carmona, S. B., Bolívar, F., Escalante, A.. Current perspectives on applications of shikimic and aminoshikimic acids in pharmaceutical chemistry. 2014. Research and Reports in Medicinal Chemistry. 4: 35–46. DOI: http://dx.doi.org/10.2147/RRMC.S46560. 4. Riveros-Mckay, F., Campos, I., Giles-Gómez, M., Bolívar, F., Escalante, A. 2014. Draft genome sequence of Leuconostoc mesenteroides P45 isolated from pulque, a traditional Mexican alcoholic fermented beverage. Genome Announcements. 2(6):e01130-14. doi:10.1128/genomeA.01130-14. 5. Torres-Rodríguez, I., Rodríguez-Alegría, Miranda-Molina, A., Giles-Gómez, M., Conca Morales, R., López-Munguía, Bolívar, F., Escalante, A. 2014. Screening and characterization of extracellular polysaccharides produced by Leuconostoc kimchii isolated from traditional fermented pulque beverage. SpringerPlus. 3:583. DOI: 10.1186/2193-1801-3-583. 6. Martínez-Rodríguez, J. C., De la Mora-Amutio, M., Plascencia-Correa, L. A., AudeloRegalado, E., Torres-Guardado, F. R., Hernández-Sánchez, E., Peña-Ramírez, Y. J. Escalante, A., Beltrán-García, Tetsuya Ogura, T. 2014. Cultivable endophytic bacteria from Agave tequilana and their role as plant growth promoter. Brazilian Journal of Microbiology. 45, 4, 1333-1339. 7. Porras-Domínguez, J., Ávila-Fernández, A., Rodríguez-Alegría, M. E., Miranda-Molina, A., Escalante, A., González-Cervantes, R., Olvera, C., López-Munguía, A. 2014. Levan-type FOS production using Bacillus licheniformis. Process Biochemistry. 49: 783–790. DOI: http://dx.doi.org/doi:10.1016/j.procbio.2014.02.005. 8. Licona Cassani, C., Lara, A., Cabrera Valladares, N., Escalante, A., Hernández Chávez, G., Martinez, A., Bolivar, F., Gosset, G. 2014. Inactivation of pyruvate kinase or the phosphoenolpyruvate:sugar phosphotransferase system increases shikimic and dehydroshikimic acid yields from glucose in Bacillus subtilis. Journal of Molecular Microbiology and Biotechnology. 24:37–45. DOI: 10.1159/000355264. 9. Balderas-Hernández, V., Treviño-Quintanilla, L. G., Hernández Chávez, G., Martinez, A., Bolivar, F., Gosset, G. 2014. Catechol biosynthesis from glucose in Escherichia coli anthranilate-overproducer strains by heterologous expression of anthranilate 1,2-dioxygenase from Pseudomonas aeruginosa PAO1. Microbial Cell Factories 2014, 13:136. DOI:10.1186/s12934-014-0136-x. 10. Centeno-Leija, S., Huerta-Beristain, G., Giles-Gómez, M., Bolivar, F., Gosset, G., Martinez, A. 2014. Improving poly-3-hydroxybutyrate production in Escherichia coli by combining the increase in the NADPH pool and acetyl-CoA availability. Antonie van Leeuwenhoek.105: 687696. DOI 10.1007/s10482-014-0124-5. 11. Sabido, A., Sigala, J. C., Hernández-Chávez, G., Flores, N., Gosset, G., Bolñivra, F. 2014. Physiological and transcriptional characterization of Escherichia coli strains lacking interconversion of phosphoenolpyruvate and pyruvate when glucose and acetate are coutilized. 111: 1150–1160. DOI: 10.1002/bit.25177 12. Cortés, J. T. Flores, N. Bolívar, F., Lara, A. R., Ramírez, O. T. 2015. Physiological effects of pH gradients on Escherichia coli during plasmid production. Biotechnology and Bioengineering. First published: 8 September 2015. DOI: 10.1002/bit.25817. 13. Aguilar, C., Flores, N., Riveros-McKay, F., Sahonero-Canavesi, D., Carmona, S. B., Geiger, O., Escalante, A., Bolívar, F. Deletion of the 2-acyl-glycerophosphoethanolamine cycle improves glucose metabolism in Escherichia coli strains employed for overproduction of aromatic compounds. Microbial Cell Factories. 14:194. DOI 10.1186/s12934-015-0382-6. 14. Martínez, J. A., Bolívar, F., Escalante, A., 2015. Shikimic acid production in Escherichia coli: From classical metabolic engineering strategies to omics applied to improve its production. Frontiers in Bioengineering and Biotechnology. 3: article 145. doi:10.3389/fbioe.2015.00145. 15. Carmona, S. B., Fabián Moreno Avitia, Francisco Bolívar, Guillermo Gosset, Escalante, A. 2015. Inactivation of the PTS as a strategy to engineer the production of aromatic metabolites in Escherichia coli. Journal of Molecular Microbiology and Biotechnology. 25:194–207. DOI: 10.1159/000380854. 16. Vargas-Tah, A., Martínez, L. M., Hernández-Chávez, G., Martínez, A., Bolívar, F., Gosset, G. 2015. Production of cinnamic and p-hydroxycinnamic acid from sugar mixtures with engineered Escherichia coli. Microbial Cell Factories 14:6. doi:10.1186/s12934-014-0185-1. Alumnos Graduados: Doctorado Dra. Andrea Sabido Ramos (Sep 2014, FBZ) Dr. César Aguilar Martínez (Feb 2015, FBZ) Dr. José Alberto Rodríguez (Mzo 2015, FBZ) Maestría M. C. Christian Hannali Cuevas (Jun 2014, FBZ) M. C. Uriel Gutiérrez Gómez (Sep 2014, AEL) Licenciatura IB Arturo Iván Motes de Oca (Abr 2014, AEL) QFB Irene Cerón Martínez (Dic 2014, AEL) QA Noé Alejandro Arroyo Mozo (Jun 2015, AEL) QFB Karina Peña Martínez (Oct 2015, AEL) Divulgación: 2014 Conferencia: Organismos transgénicos, sus grandes beneficios y la ausencia de daño. Facultad de Química, UNAM. Abril de 2014 (FBZ). Conferencia: Organismos transgénicos, sus grandes beneficios y la ausencia de daño. Aula Mayor, El Colegio Nacional. México, D. F. Junio de 2014 (FBZ). Conferencia: Organismos transgénicos, sus grandes beneficios y la ausencia de daño. Auditorio de la Biblioteca Central. Biblioteca en el Campus Norte, Universidad Autónoma del Estado de Morelos. Cuernavaca, Morelos. Octubre 2014 (FBZ). Conferencia Magistral en la Ceremonia Protocolaria de Inauguración del 2° Congreso de Jóvenes Investigadores en el Estado de Querétaro. Organismos transgénicos, sus grandes beneficios y la ausencia de daño. Centro de Negocios de la UAQ. Universidad Autónoma de Querétaro. Querétaro, Qro. Noviembre 2014 (FBZ). Conferencia: Análisis de la diversidad microbiana y metabólica presente en el pulque, presentada en el Curso de Microbiología Avanzada de Alimentos de la Maestría en Ciencias de Alimentos, Instituto de Ciencias Agropecuarias, Universidad Autónoma del Estado de Hidalgo. 22 de septiembre de 2014. Tulancingo de Bravo, Hidalgo. 24 de noviembre de 2014 (AEL). Conferencia: Análisis de la diversidad microbiana y metabólica presente en el pulque, presentada como seminario del Grupo de Investigación en Tecnologías del Frío. Escuela Superior de Apan, Universidad Autónoma del Estado de Hidalgo. 22 de septiembre de 2014. Conferencia: Ingeniería de vías metabólicas en cepas de Escherichia coli PTS- para la producción de shikimato, presentada como Seminario del Departamento de Bioquímica, Facultad de Química, UNAM. 14 de Febrero de 2014. Plática: Aspectos generales del manejo de microorganismos genéticamente modificados, presentada durante el Primer Curso de Bioseguridad dirigido a estudiantes y personal académico del IBT-UNAM. Instituto de Biotecnología, UNAM. 23 de Enero de 2014. 2015 Conferencia: Biotecnología: Organismos transgénicos, sus grandes beneficios y la ausencia de daño. Aula Mayor. El Colegio Nacional. México, D.F. Marzo 2015 (FBZ). Conferencia: Biotecnología: Organismos transgénicos, sus grandes beneficios y la ausencia de daño. Auditorio A. facultad de Química, UNAM. México, D.F. Abril de 2015 (FBZ). Conferencia: Biotecnología: Organismos transgénicos, sus grandes beneficios y la ausencia de daño. Aula Mayor. El Colegio Nacional. México, D.F. Junio de 2015 (FBZ). Conferencia: Biotecnología: Organismos transgénicos, sus grandes beneficios y la ausencia de daño. Aula Mayor. El Colegio Nacional. México, D.F. Junio de 2015 (FBZ). Conferencia: Biotecnología: Organismos transgénicos, sus grandes beneficios y la ausencia de daño. Auditorio Arq. Pedro Ramírez Vázquez. Rectoría General de la Universidad. Autónoma Metropolitana. México, D.F. Julio 2015 (FBZ). Conferencia: Las bacterias del pulque. Conferencia presentada en la en la VI Semana de Procariontes del 24 al 26 de noviembre de 2015. Facultad de Ciencias, UNAM. México, Distrito Federal, 26 de Noviembre de 2015 (AEL). Conferencia: Análisis de la diversidad bacteriana presente en el pulque, presentada en la VI Jornada de Ingeniería en Alimentos, Universidad Tecnológica de la Mixteca. Huajuapan de León, Oaxaca, 16 de Octubre de 2015 (AEL). Conferencia: Ingeniería de vías metabólicas en bacterias para la producción de compuestos aromáticos de interés industrial, presentada en la 6ta Expo 2015 Quimilab. Guadalajara, Jalisco, 29 de mayo de 2015 (AEL). Plática: Trasncriptómica y metagenomas, presentada en el Curso de Métodos fisicoquímicos aplicados a las Ciencias Biomédicas, impartido en la Maestría en Ciencias Biomédicas, Universidad Autónoma del Estado de Guerrero. Chilpancingo, Guerrero, 19 de marzo de 2015 (AEL). Conferencia: Potencial probiótico de las bacterias lácticas del puque, presentada en la Facultad de Química, Universidad Nacional Autónoma de México y organizada por la Sociedad Estudiantil de Químicos Farmacéuticos Biólogos el 13 de Marzo de 2015 (AEL). Donativos: 1. Ingeniería Celular y Biocatálisis 740 2. CONACYT Ciencia Básica proyecto 240519 3. PAPIIT IN207914 Participación en docencia: Curso de Fisiología Microbiana, Carrera de Química Farmacéutico Biológica, Facultad de Química, UNAM. Semestres 2014-2, 2015-1/2, 2016-1. Curso de Ingeniería de Vías Metabólicas, Posgrado en Ciencias Bioquímicas – UNAM. Instituto de Biotecnología, UNAM. Semestres 2015-1, 2016-1. Títulos y autores de los carteles que presentará el grupo: 1. Estudio del impacto de las mutaciones desarrolladas durante un proceso de evolución adaptativa de la cepa de Escherichia coli PB11 para mejorar su capacidad de crecimiento en glucosa. Susy Beatriz Carmona, Adelfo Escalante, Ramón de Anda, Francisco Bolívar. 2. Ingeniería de proteínas para la obtención de una shikimato deshidrogenasa con menor inhibición por shikimato y mayor eficiencia catalítica. Dulce Díaz Quiroz, Francisco Bolívar, Adelfo Escalante. 3. Aislamiento, tipificación e identificación de bacterias ácido lácticas productoras de exopolisacáridos en muestras de aguamiel y pulque. David López Soto, Francisco Bolívar, Adelfo Escalante. 4. Efecto de la inactivación de las enzimas shikimato cinasas sobre el crecimiento y la producción de shikimato en medio mínimo en una cepa de Escherichia coli PTS-. Fabián Moreno Avitia, Adelfo Escalante, Ramón de Anda, Francisco Bolívar. Grupo de Jean Louis Charli (2014-2015) Título: Una ventana al control central del balance de energía Resumen: Nuestro cerebro tiene un papel fundamental en el mantenimiento del balance de energía, o sea controla los procesos de ingesta de alimento, distribución de los substratos energéticos, y gasto energético (metabolismo basal, termogénesis, actividad física…). Para este propósito, integra informaciones internas y externas y genera respuestas comportamentales y metabólicas apropiadas. Una diversidad de circuitos neurales distribuidos a todo lo largo del cerebro participa en estas respuestas. Uno de los nodos de estos circuitos es una región localizada a la base del cerebro llamada hipotálamo. El hipotálamo recibe directamente o indirectamente información proveniente de la periferia relacionada a la ingesta y a las reservas de energía; esta información es retransmitida localmente y a fuera del hipotálamo para el control de la ingesta; por otro lado, salidas autónomas y neuroendocrinas controlan el gasto energético. Las neuronas hipotalámicas implicadas en estos procesos son numerosas y tienen fenotipos diversos. Uno de estos fenotipos es la capacidad de sintetizar y secretar la hormona liberadora de tirotropina (TRH), un tripéptido que en los mamíferos es sintetizado por neuronas del núcleo paraventricular (NPV) del hipotálamo. Del NPV la TRH es transportada a la eminencia media (EM) para liberarse al sistema portal que irriga a la adenohipófisis, donde controla la secreción de la tirotropina, la hormona encargada del control de la secreción de las hormonas tiroideas por la glándula tiroides. Este eje controla por lo tanto el gasto metabólico celular y la termogénesis facultativa. Las señales de tipo peptidergico que las neuronas emiten son controladas a múltiples niveles. Uno de los mecanismos principales de inactivación de péptidos es la actividad de peptidasas embebidas en la membrana plasmática, con su sitio activo en el lado externo de la membrana (ectoenzimas). Nuestro laboratorio identificó una ectopeptidasa específica para la TRH, la piroglutamato peptidasa II (PPII). Actualmente, intentamos determinar cuál es el significado fisiológico de la presencia de esta ectoenzima en tanicitos de la EM, células gliales que forman la pared del tercer ventrículo, con extensiones en íntimo contacto con las terminales nerviosas TRHérgicas. En este sitio la PPII parece controlar la cantidad de TRH que estimula la secreción de tirotropina. Para confirmar esta hipótesis, estamos analizando el efecto de retos a la homeostasis energética sobre la expresión de la PPII en tanicitos, e intentamos desarrollar herramientas para manipular la expresión de la PPII específicamente en los tanicitos. Finalmente, en ratones nulos para la PPII estudiamos el estado del eje hipotálamo-hipófisis-tiroidea y del balance de energía. Neuronas que expresan a la TRH en otras regiones hipotalámicas tienen un papel menos claro que las neuronas del NPV, si bien se sospecha que algunas tienen un papel relevante en el control del balance de energía. Uno de nuestros propósitos es identificar las neuronas TRHergicas hipotalámicas involucradas durante la inducción de una obesidad experimental, y definir cuál es su contribución al fenotipo. Por otro lado, la TRH ejerce un potente efecto anoréxico (suprime la ingesta de alimentos) in vivo independiente del control del eje tiroideo, efecto que se observa cuando se inyecta la TRH en el hipotálamo. Así, en otro proyecto, intentamos determinar si las neuronas TRHergicas del núcleo dorsomedial del hipotálamo transmiten una señal anoréxica hacia el hipotálamo lateral, una región que controla la ingesta de alimentos. Integrantes del Grupo: Rosa María Uribe Antonieta Cote Miguel Cisneros Ivan Lazcano Adair Rodríguez Yamili Vargas Melissa Rosas Esau Urbina Edgar García Ricardo Ayala Marlen Ramírez Beetsi Urrieta Laura Urzua Oscar Bastida Publicaciones: 1. R.M. Uribe, L. Jaimes-Hoy, C. Ramírez-Martínez, A. García-Vázquez, F. Romero, M. Cisneros, A. Cote-Vélez, J.L. Charli, P. Joseph-Bravo. 2014. Voluntary exercise adapts the hypothalamus pituitary thyroid axis in male rats. Endocrinology, 155, 2020-2030. 2. V. Rodríguez-Molina, J. Patiño, Y. Vargas, E. Sánchez-Jaramillo, P. Joseph-Bravo, J.-L. Charli. 2014.TRH regulates action potential shape in cerebral cortex pyramidal neurons. Brain Research, 1571, 1-11. 3. Y. Méndez, K. Pérez-Labrada, J. González-Bacerio, G. Valdés, M. Á. Chávez, J. Osuna, J.-L. Charli, I. Pascual, D.G. Rivera. 2014. Combinatorial multicomponent access to natural-productsinspired peptidomimetics: Discovery of selective inhibitors of microbial metalloaminopeptidases. ChemMedChem, 9, 2351-2359. 4. J. González-Bacerio, J. Osuna, A. Ponce, R. Fando, K. Figarella, Y. Méndez, J.-L. Charli, M.Á. Chávez. High-level expression in Escherichia coli, purification and kinetic characterization of Plasmodium falciparum M1-aminopeptidase. Protein Expression and Purification, 104, 103114. 5. I. Sotelo-Rivera, L. Jaimes-Hoy, A. Cote-Vélez, C. Espinosa-Ayala, J.-L. Charli, P. JosephBravo. 2014. An acute injection of corticosterone increases thyrotrophin releasing hormone expression in the paraventricular nucleus of the hypothalamus but interferes with the rapid hypothalamus pituitary thyroid axis response to cold in male rats. Journal of Neuroendocrinology, 26, 882–890. 6. J. González-Bacerio, R. Fando, A. del Monte-Martínez, J.-L. Charli, M. de los Á. Chávez. 2014. Plasmodium falciparum M1-aminopeptidase: a promising target for the development of antimalarials. Current Drug Targets, 15, 1144-1165. 7. M. Martínez-Armenta, S. Díaz de León-Guerrero, A. Catalán, L Alvarez-Arellano, R.M. Uribe, M. Subramaniam, J.-L. Charli, L. Pérez-Martínez. 2015. TGFβ2 regulates hypothalamic Trh expression through the TGFβ inducible early gene-1 (TIEG1) during fetal development. Molecular and Cellular Endocrinology, 400, 129-139. 8. P. Joseph-Bravo, L. Jaimes-Hoy, J.-L. Charli. 2015. Regulation of TRH neurons and energy heomeostasis related signals under stress. Journal of Endocrinology, 224, R139-R159. 9. I. Lazcano, A. Cabral, R. M. Uribe, L. Jaimes-Hoy, M. Perello, P. Joseph-Bravo, E. Sánchez J.-L. Charli. 2015. Fasting enhances pyroglutamyl peptidase II activity in tanycytes of the mediobasal hypothalamus of male adult rats. Endocrinology, 156: 2713–2723. 10. P. Joseph-Bravo, L. Jaimes-Hoy, R.M. Uribe, J-L. Charli. 2015. TRH, the first hypophysiotropic releasing hormone isolated: control of pituitary-thyroid axis. Journal of Endocrinology, 226: 85-100. 11. E.N. Jiménez-Vázquez, C.E. Díaz-Velásquez, R.M. Uribe, J.M. Arias, U. García. 2015. Molecular cloning and expression of a GABA receptor subunit from the crayfish Procambarus clarkii. Journal of Neuroscience Research. doi: 10.1002/jnr.23695. 12. R. M. Uribe, V. Trujillo-Alonso, A. Cote-Vélez, P. Joseph-Bravo, J.-L. Charli. Noradrenaline enhances TRH mRNA levels in the paraventricular nucleus of the hypothalamus. Sometido. 13. A. Cote-Vélez, A. Martínez Báez, R. M. Uribe, P. Joseph-Bravo and J.-L. Charli. A screen for modulators of TRH expression in hypothalamic cell culture reveals that orexin A and B have rapid and opposite effects on TRH expression and release. Sometido. Alumnos Graduados: María del Pilar Torres Reyes. Licenciatura en Biología, Facultad de Ciencias, UNAM. Junio 2014. (RMU) Yamili Vargas Orihuela. Maestría en Ciencias Bioquímicas, UNAM. Diciembre 2014. (JLC) Ivan Lazcano Sánchez. Doctorado en Ciencias Biológicas, UNAM. Julio 2015. (JLC) Edgar García Palacios. Licenciatura en Biología, UAEM. Agosto 2015 (AC) Ricardo Ayala Uribe. Licenciatura en Biología, UAEM. Noviembre 2015 (AC) Donativos: 1. CONACYT (Ciencia básica) convenio 154931. 2012-2014. (JLC) 2. CONACYT (Ciencia básica) convenio 128665. 2012-2015 (RMU) 3. Dirección General de Asuntos del Personal Académico de la UNAM: (PAPITT IN206712”). 2012-2014. (JLC) 4. Dirección General de Asuntos del Personal Académico de la UNAM. (PAPITT IN208515). 2015-2017. (JLC) Participación en docencia: Participación en el Taller “La Biología a partir de las biomoleculas: nuevos paradigmas y aplicaciones”, Facultad de Ciencias, UNAM: varias clases cada semestre en 2015 (JLC, RMU) Títulos y autores de los cárteles que presentará el grupo: 1. Análisis de la expresión de la piroglutamil peptidasa II en cultivos de tanicitos. Adair Rodríguez, Antonieta Cote-Vélez, Beetsi Urrieta, Patricia Joseph y Jean-Louis Charli. 2. Análisis del fenotipo de ratones nulos para la piroglutamil peptidasa II. Antonieta Cote-Vélez, Ricardo Ayala, Edgar García, Patricia Joseph y Jean-Louis Charli. 3. ¿La proyección TRHergica entre el núcleo dorsomedial del hipotálamo y el hipotálamo lateral transmite una señal anoréxica? Yamili Vargas, Esau Urbina, Rosa María Uribe, Edith Sánchez, Patricia Joseph y Jean-Louis Charli. Grupo de Enrique Rudiño Piñera (2014-2015) Título: Usando las limitaciones de la difracción de rayos X para describir procesos biológicos” Resumen: Las oxidasas multicobre (MCOs por sus siglas en inglés) son enzimas que contienen iones cobre, los cuales tienen un papel central en la catálisis de estas enzimas, interviniendo en la oxidación de diversos sustratos y generando una transferencia de electrones a un átomo de oxígeno para su conversión en agua con la adición de protones. Todo esto bajo un mecanismo de reacción catalítica, que si bien ha sido profusamente estudiado en diversos sistemas, aún no se encuentra caracterizado en su totalidad. Adicionalmente, es muy común que las estructuras cristalográficas de las MCOs resulten en la pérdida, parcial o total, de ocupación en uno o varios de los iones cobre. En nuestro grupo de trabajo se ha determinado la estructura cristalográfica de la oxidasa multicobre de Thermus thermophilus (MCO-Tth) (código PDB 2XU9). Sin embargo, la exposición a los rayos X produce modificaciones específicas en la estructura: la ocupación de uno de los cuatro cobres catalíticos en esta metaloenzima, el CuT2, está particularmente afectada, inclusive en dosis de radiación absorbida muy bajas (0.7 MGy). Por lo tanto, en uno de los proyectos realizados en nuestro grupo, exploramos distintas condiciones como el uso de moléculas radioprotectoras, el uso de líneas de alta energía en fuentes sincrotrónicas, así como la atenuación en líneas estándar del flujo de rayos X. Todo lo anterior con el fin último de preservar la ocupación de los cobres en estudios cristalográficos en MCOs, particularmente del CuT2 en los cristales de la MCO-Tth. En este contexto es importante recordar que la catálisis en metaloenzimas depende de la preservación de los metales, y al escindirlos durante los experimentos de difracción de rayos X, pueden producirse cambios grandes o sutiles en la estructura que pueden conducir a conclusiones biológicas equivocadas, o bien producir cambios que permitan elucidar los procesos redox que también ocurrirían en el funcionamiento biológico de estas enzimas. Una vez conseguido este objetivo utilizamos a las estructuras generadas para intentar comprender de manera más amplia el mecanismo catalítico de este grupo de enzimas. Nuestros resultados muestran la complejidad del sistema, sin embargo, también muestra la viabilidad de extraer información sobre dinámica molecular y flujos de electrones y protones en proteínas redox empleado técnicas de difracción de rayos X. -Integrantes del Grupo 2014-2015 Enrique Rudiño Piñera, Claudia Rodríguez Almazán, Sonia Patricia Rojas Trejo, César Salvador Cardona Félix, Adelaida Díaz Vilchis, Rayana Ramona Ruiz Arellano, Víctor Rivelino Juárez González, Hugo Javier Serrano Posada, Ulises Salazar Kuri, Andrés Zarate Romero, Adam Andrés Campos Acevedo, Yasel Guerra Borrego, Ariadna Berenice Juárez Martínez, Jesús Lara Popoca, Rubén Priego Jiménez, Santos Ramírez Carreto, Nizaa Jiménez Arrollo, Francisco Murphy Pérez, Arisbeth Guadalupe Almeida Juárez, Osvaldo Gómez Secundino, Beatriz Miranda Zaragoza, Alberto Antony Venancio Landeros, Edith Flores Hernández, Víctor Hugo Cifuentes Castro, Elena Lizbeth García Villegas, Francisco Cortes Trujillo, Juan José Cárdenas Solano, Fátima Ugalde Mercado, Idania Emedith Quintero Reyes, Graciela Espinoza Luna, Edith Alanís Rojas, Lesly Jayuri Tapia Arriaga, José Antonio Gallardo Lozada, Ángel Eduardo Fuentes Martínez, Sigrid Yunuen Mares Tapanohaya, Julián González Borrayo, Dalia Betancourt Tabaco, Aurelia Ocampo Vargas, María De la Paz Colín Romero, Mariana Ortiz Ramos -Publicaciones 2014-2015 1. Serrano-Posada,H. Centeno-Leija,S. Rojas-Trejo,S.P. Rodriguez-Almazan,C. Stojanoff,V. Rudino-Pinera,E. 2015. X-ray-induced catalytic active-site reduction of a multicopper oxidase: structural insights into the proton-relay mechanism and O2reduction states Acta Crystallographica Section D,71, 2396-2411. 2. Riano-Umbarilla,L. Ledezma-Candanoza,L.M. Serrano-Posada,H. FernandezTaboada,G. Olamendi-Portugal,T. Rojas-Trejo,S. Gomez-Ramirez,I.V.Rudino-Pinera,E. Possani,L.D. Becerril,B. 2015. Optimal neutralization of Centruroides noxius venom is understood through a structural complex between two antibody fragments and the Cn2 toxin Journal of Biological Chemistry, Nov 20 [Epub ahead of print], . 3. Serrano-Posada,H. Centeno-Leija,S. Rojas-Trejo,S. Stojanoff,V. Rodriguez-Sanoja,R. Rudino-Pinera,E. Sanchez,S. 2015. Crystallization and X-ray diffraction analysis of a putative bacterial class I labdane-related diterpene synthase Acta Crystallographica Section F: Structural Biology and Crystallization Communications, 71, 1194-1199. 4. Bertrand,B. Martinez-Morales,F. Tinoco-Valencia,R. Rojas,S. Acosta-Urdapilleta,L. Trejo-Hernandez,M.R. 2015. Biochemical and molecular characterization of laccase isoforms produced by the white-rot fungus Trametes versicolor under submerged culture conditions Journal of Molecular Catalysis B: Enzymatic, 122, 339-347. 5. Villalba,M.I. Canul-Tec,J.C. Luna-Martinez,O.D. Sanchez-Alcala,R. OlamendiPortugal,T. Rudino-Pinera,E. Rojas,S. Sanchez-Lopez,R. Fernandez-Velasco,D.A. Becerril,B. 2015. Site-directed Mutagenesis Reveals Regions Implicated in the Stability and Fiber Formation of Human lambda3r Light Chains Journal of Biological Chemistry, 290, 2577-2592 [Correction vol 290 p 13039]. 6. Campos-Acevedo,A.A. Rudino-Pinera,E. 2014. Crystallographic Studies Evidencing the High Energy Tolerance to Disrupting the Interface Disulfide Bond of Thioredoxin 1 from White Leg Shrimp Litopenaeus vannamei Molecules, 19, 21113-21126. 7. Lopez-Falcon,B. Meyer-Nava,S. Hernandez-Rodriguez,B. Campos,A. Montero,D. Rudino,E. Vazquez,M. Zurita,M. Valadez-Graham,V. 2014.Characterization of the Drosophila Group Ortholog to the Amino-Terminus of the Alpha-Thalassemia and Mental Retardation X-Linked (ATRX) Vertebrate Protein PLoS ONE, 9, e113182. 8. Ghaffari,N. Sanchez-Flores,A. Doan,R. Garcia-Orozco,K.D. Chen,P.L. OchoaLeyva,A. Lopez-Zavala,A.A. Carrasco,J.S. Hong,C. Brieba,L.G. Rudino-Pinera,E. Blood,P.D. Sawyer,J.E. Johnson,C.D. Dindot,S.V. Sotelo-Mundo,R.R. Criscitiello,M.F. 2014. Novel transcriptome assembly and improved annotation of the whiteleg shrimp (Litopenaeus vannamei), a dominant crustacean in global seafood mariculture Scientific Reports, 4, 7081. 9. Lopez-Zavala,A.A. Quintero-Reyes,I.E. Carrasco-Miranda,J.S. Stojanoff,V. Weichsel,A. Rudino-Pinera,E. Sotelo-Mundo,R.R. 2014. Structure of nucleoside diphosphate kinase from pacific shrimp (Litopenaeus vannamei) in binary complexes with purine and pyrimidine nucleoside diphosphatesActa Crystallographica Section F: Structural Biology and Crystallization Communications, 70, 1150-1154. 10. Bertrand,B. Martinez-Morales,F. Tinoco,R. Rojas-Trejo,S. Serrano-Carreon,L. Trejo- Hernandez,M.R. 2014. Induction of laccases in Trametes versicolor by aqueous wood extracts World Journal Of Microbiology & Biotechnology, 30, 135-142. 11. Carrasco-Miranda,J.S. Lopez-Zavala,A.A. Arvizu-Flores,A.A. Garcia-Orozco,K.D. Stojanoff,V. Rudino-Pinera,E. Brieba,L.G. Sotelo-Mundo,R.R. 2014.Crystal Structure of the Shrimp Proliferating Cell Nuclear Antigen: Structural Complementarity with WSSV DNA Polymerase PIP-Box PLoS ONE, 9, e94369. 12. Bello,M. Correa-Basurto,J. Rudino-Pinera,E. 2014. Simulation of the cavity-binding site of three bacterial multicopper oxidases upon complex stabilization: interactional profile and electron transference pathways Journal of Biomolecular Structure and Dynamics, 32, 1303-1317. -Alumnos Graduados 2014-2015 Licenciatura: Estefanía Jaramillo Bustamante, “Clonación y expresión del antígeno nuclear de proliferación celular (PCNA) del hipertermófilo Thermococcus gammatolerans”, 4-032014, Ingeniería en Biotecnología. Juan José Cárdenas Solano, “Cristalización de la Metacaspasa tipo II de Nicotiana tabacum. Expresión, Purificación y cristalización del anticuerpo 202F ScvF de Humano”, 11-04-2014, Ingeniería en Biotecnología. Elena Lizbeth García Villegas, “Cristalización de la catalasa-peroxidasa de Neurospora crassa”, 20-11-2015, Licenciado en Biología. -Alumnos en Proceso Doctorado Rubén Priego Jiménez Adam Andrés Campos Acevedo Jesús Lara Popoca Yasel Guerra Borrego Ariadna Berenice Juárez Martínez Santos Ramírez Carreto Maestría Francisco Murphy Pérez Osvaldo Gómez Secundino Alberto Antony Venancio Landeros Arisbeth Guadalupe Almeida Juárez Víctor Hugo Cifuentes Castro Nizaa Jiménez Arroyo Beatriz Miranda Zaragoza Edith Flores Hernández Elena Lizbeth García Villegas Licenciatura Francisco Cortes Trujillo Dalia Betancourt Tabaco -Donativos 2014-2015 1. PAPIIT-UNAM IN109114, $600,000 pesos en tres años, inicio 2014. 2. CONACYT CIENCIA BASICA, $1,350,000 pesos en tres años, inicio 2013. -Títulos y autores de los cárteles que presentará el grupo. Yasel Guerra Borrego "Structural and kinetic studies of the inhibition mechanisms of bifunctional STI-Kunitz protease inhibitors on aspartic proteases". Adam Andrés Campos Acevedo “Analysis of the dimeric interface of thioredoxin 1 from white leg shrimp Litopenaeus vannamei and the effects of mutation on structure.” Grupo de Hilda Lomeli (2014-2015) Título: El sistema CRISPR/Cas para el estudio del desarrollo embrionario del pez cebra: mutaciones en el gen baf57 causan defectos en el corazón Resumen: En nuestro laboratorio utilizamos el pez cebra para abordar el estudio de la función de genes que participan en el desarrollo embrionario. Hemos logrado inactivar la expresión genética a través de distintas estrategias. En los últimos dos años dedicamos parte de nuestro esfuerzo a implementar el sistema CRISPR/Cas para introducir mutaciones específicas en el genoma de pez. A la fecha, ya hemos introducido mutaciones heredables en varios genes. En la presentación describiré nuestros avances en este tema. Una de las líneas transgénicas que generamos tiene mutaciones en el gen baf57, el cual codifica para una subunidad que forma parte del complejo remodelador de cromatina SWI/SNF. El complejo SWI/SNF (o BAF en vertebrados) está presente en todas las células de eucariotes y actúa a través de su actividad de ATPasa para modificar la estructura de la cromatina y así facilitar la unión de factores proteicos al DNA. Un gran número de genes involucrados en procesos celulares, tales como la diferenciación, el ciclo celular y el mantenimiento de células troncales, son transcripcionalmente controlados por el complejo SWI/SNF. BAF57 es una subunidad que siempre forma parte del complejo SWI/SNF, independientemente de la combinatoria de otras subunidades. Esta y otras de sus propiedades evolutivas y estructurales sugieren que BAF57 podría tener un papel esencial para el funcionamiento de SWI/SNF en vivo. En la presentación se mostrarán evidencias obtenidas del estudio de la línea mutante, que confirman que BAF57 tiene una participación esencial durante el desarrollo embrionario, y en particular, es requerida durante la cardiogénesis para la diferenciación del atrio y el ventrículo. También se hará mención, brevemente, de nuestros avances en otros proyectos. Integrantes del Grupo: Investigadores Hilda Lomelí Buyoli Enrique Salas Vidal Denhí Schnabel (colaboradora), Técnicos Laura Ramírez Estudiantes Jorge Castillo Robles (doctorado-H.Lomelí) Francisco Castillo Castellanos (doctorado-H.Lomelí) Roberto Moreno Ayala (doctorado-H.Lomelí) Mario Mendieta Serrano (doctorado-E.Salas) Jerónimo Miranda Rodríguez (doctorado-D.Schnabel) Javier Méndez Cruz (maestría-E.Salas). Personal Administrativo Dulce Pacheco Benitez Minerva Carcaño Virginia Ramírez Granados Publicaciones: 1. Moreno-Ayala R, Schnabel D, Salas-Vidal E, Lomelí H. 2015. PIAS-like protein Zimp7 is required for the restriction of the zebrafish organizer and mesoderm development. Developmental Biology. 403(1):89-100 2. Mendieta-Serrano MA, Schnabel D, Lomelí H, Salas-Vidal E. 2015. Spatial and temporal expression of zebrafish glutathione peroxidase 4 a and b genes during early embryo development. Gene Expr Patterns. 19(1):98-107 Alumnos graduados: Roberto Moreno (HL)- Doctorado (2015) Francisco Castillo (HL)- Maestría (2014) Javier Méndez Cruz (ES)- Licenciatura (2015) Donativos: 1. CONACYT 2015- Abril 2017 2. DGAPA 2014-2016 Participación en docencia: Participante en el curso de Biología Celular semestres 2014-2 y 2015-2. Participante en el curso de Biología Molecular de posgrado en UAEM Divulgación: 1. Participación en un ejercicio de divulgación titulado “Diálogos en la Ciencia” dentro del proyecto “Divulgación de Temas de Ciencia Contemporánea” organizado por la DGDCCCADET/ UNAM. Dentro de esta actividad, que tuvo lugar en el 2014, se produjo un video que contiene una entrevista en la que describo mi trabajo y que puede verse en línea. 2. Día de puertas abiertas, mayo 2014 (todo el grupo) 3. Participación en una entrevista por parte de la Lic. Carmen Baez para la Agencia Informativa de CONACYT. Con base en ella, la Lic. Báez escribió un artículo de divulgación, junio 2015. 4. Participación en entrevista para el Instituto Morelense de Radio y Televisión (IMRyTv) que salió al aire el martes 11 de agosto del 2015. Títulos y autores de los carteles que presentará el grupo: 1. Papel de la Rho-GTPasa RhoA en la segregación del plasma germinal en el pez cebra. Jerónimo Miranda, Denhí Schnabel. 2. Participación de especies de oxígeno reactivas derivadas de la NADPH-oxidasa en la migración por epibolia del embrión de pez cebra. Mario Mendieta, Javier Méndez, Enrique Salas. 3. Determinación de la función del gen zimp10 en el desarrollo del pez cebra Francisco Castillo. Participantes Miercoles 9 de diciembre del 2015 Grupo de Joseph Dubrovsky (2014-2015) Título: Morfogénesis de la raíz lateral antes de su nacimiento Resumen: La formación de los sistemas radicales y su desarrollo dependen del crecimiento de la raíz y su ramificación. Ambos procesos están relacionados a la formación y mantenimiento del Meristemo Apical de la Raíz (MAR). En nuestro laboratorio estamos interesados en entender cómo se forman los nuevos MARs de las raíces laterales (RLs) y cómo el MAR mantiene o pierde su actividad. Previamente demostramos que muchas de las especies de la familia Cactaceae exhiben agotamiento del MAR. Para entender cómo ocurre ésto hemos ensamblado de novo el transcriptoma de Pachycereus pringlei, un cactus columnar endémico de México, e identificamos a los genes expresados diferencialmente (ver el cartel Presentado por el estudiante de Doctorado, M. en C. Gustavo Rodríguez-Alonso). También hemos aislado a mutantes de Arabidopsis thaliana cuya arquitectura del sistema radical está afectada, y mediante secuenciación masiva del ADN de población de mapeo, identificamos a los genes responsables de los fenotipos alterados. Algunas de las mutantes que hemos aislado exhiben anomalías en el desarrollo de las RLs y representan una buena herramienta para estudiar la morfogénesis de las mismas. El enfoque principal de la presentación será el análisis de la morfogénesis de los primordios de las raíces laterales (PRLs). Durante mi año sabático, realizado en la Universidad de Oxford, fue empleado el análisis clonal del desarrollo de los PRLs de Arabidopsis usando el sistema de transposición de elementos Ds por la transposasa Ac del maíz. Este análisis clonal demostró que los escenarios de la iniciación de los PRLs son más diversos de lo que anteriormente se conocía, lo cual indica que el proceso de la iniciación de los PRLs es bastante flexible. Otra parte de mi plática será dedicada al análisis de la morfogénesis del PRL en las mutantes bajo estudio en nuestro laboratorio. Un fenotipo más notorio en el desarrollo de los PRLs es en las mutantes recesivas en el gen ARABIDOPSIS HOMOLOG of TRITHORAX1 (ATX1). ATX 1 codifica para una metiltransferasa de H3K4 y la mutante atx1-1 forma PRLs asimétricos. Para identificar a los genes blanco regulados por ATX1 obtuvimos datos de RNA-seq de la raíz de una de las mutantes en ATX1 (ver el cartel presentado por M. en C. Selene Napsucialy Mendivil). Otras mutantes que estudiamos en el laboratorio también presentan varias anomalías en la formación de patrón durante el desarrollo del PRL y estos datos serán presentados. La investigación realizada en nuestro laboratorio es multidisciplinaria e involucra el uso de técnicas de microscopía confocal, análisis de imágenes y análisis de tipo “time-lapse”, que en conjunto con una amplia gama de marcadores moleculares, nos ayudan a entender cómo ocurre la morfogénesis de los PRLs y cuáles son los mecanismos moleculares y celulares involucrados en su desarrollo. Integrantes del Grupo: Investigadores Dra. Svetlana Shishkova Técnicos M. en C. Selene Napsucialy Mendivil I.B.I. Marcela Ramírez Yarza Estudiantes de Servicio Social y Estancias en el Laboratorio Alexis O. Campuzano Glez. (Universidad Politécnica de Morelos). Duración de 4 meses. Jocelyn Aurelia Briones Robles (Universidad Politécnica de Morelos). Duración de 6 meses. Sinhue Raphael Albarrán Hernández (Escuela de Técnicos Laboratoristas/UAEM). Duración de 6 meses. María de la Luz Moreno (Escuela de Técnicos Laboratoristas/UAEM). Duración de 6 meses. Javier Yael Solis (Escuela de Técnicos Laboratoristas/UAEM). Duración de 6 meses. Daniela Fernanda Pecina Ornelas (Escuela de Técnicos Laboratoristas/UAEM). Duración de 6 meses. Angélica Contreras Valladares (Escuela de Técnicos Laboratoristas/UAEM). Duración de 6 meses. Yaritza Vergara Ortiz (Universidad Politécnica de Morelos). Duración de 4 meses. Biol. Aranza Xhaly Quintana Armas (Universidad Autónoma Metropolitana, Unidad Iztapalapa). Estancia de 7 meses. Estudiantes de Licenciatura Carlos Alfonso Sierra Sarabia Jessica Pacheco Mendoza Alberto Hernández Juarez Alexis O. Campuzano Glez. Estudiantes de Maestría Lic. Mayra Liliana Lopez Valle Biol. Hector Hugo Torres Martínez Estudiantes de Doctorado Lic. Andrés Cuevas Moreira M. en C. Gustavo Rodríguez Alonso Biol. Blanca Jazmín Reyes Hernández M. en C. Ramcés De Jesús García M. en C. Selene Napsucialy Mendivil M. en C. Hector Hugo Torres Martínez Personal Administrativo Jesús Moreno Mercado Margarita Ferrel Fuentes Juana Maricela Izquierdo Cabrera Publicaciones: 1. López-Bucio J.S., Dubrovsky J. G., Raya-González J., Ugartechea-Chirino Y., López-Bucio J., de Luna-Valdez L.A., Ramos-Vega M., León P., Guevara-García A.A. 2014. Arabidopsis thaliana mitogen-activated protein kinase 6 is involved in seed formation and modulation of primary and lateral root development. Journal of Experimental Botany, 65: 169-183, doi:10.1093/jxb/ert368 IF 5.794 2. Reyes-Hernández B.J., Srivastava A.C., Ugartechea-Chirino Y. Shishkova S., Ramos-Parra P.A., Lira-Ruan V., Díaz de la Garza R.I., Dong G., Moon J.-C., Blancaflor E.B., Dubrovsky J.G. 2014. Root indeterminacy-to-determinacy developmental switch is operated through a folate dependent pathway in Arabidopsis thaliana. New Phytologist, 202: 1223–1236, doi: 10.1111/nph.12757 IF 6. 545 3. Napsucialy-Mendivil S., Alvarez-Venegas R., Shishkova S., Dubrovsky J.G. 2014. ARABIDOPSIS HOMOLOG of TRITHORAX1 (ATX1) is required for cell production, patterning, and morphogenesis in root development. Journal of Experimental Botany, 65: 6373-6384, doi: 10.1093/jxb/eru355 IF 5.794 4. Dubrovsky J.G., Barlow P.W. 2015. The origins of the quiescent centre concept, New Phytologist, 206: 493–496, DOI: 10.1111/nph.13307, IF 7.672 5. Ivanchenko M., Zhu J., Wang B., Medvecka E., Du Y., Azzarello E., Mancuso S., Megraw M., Filichkin S., Dubrovsky J. G., Friml J., Geisler M. 2015. The cyclophilin-A DIAGEOTROPICA gene affects auxin transport in both root and shoot to control lateral root formation. Development, 142: 1-10, doi:10.1242/dev.113225, IF 6.462 6. Medina-Andrés R., Solano-Peralta A., Saucedo-Vázquez J.P., Napsucialy-Mendivil S., Pimentel-Cabrera J.A., Sosa-Torres M.A., Dubrovsky J.G., Lira-Ruan V. 2015. The nitric oxide production in the moss Physcomitrella patens is mediated by nitrate reductase. PLOS ONE 10(3): e0119400. doi:10.1371/journal.pone.0119400 IF 3.234 Capitulos de libros: 6. Dubrovsky J.G. and Laskowski M. 2015. Root development: Lateral root initiation. In: Encyclopedia of Applied Plant Sciences, 2nd Ed. Brian Thomas, Denis J. Murphy, Brian G. Murray (eds.). Oxford, UK: Elsevier Academic Press (ISBN: 0122270509) En prensa. Alumnos graduados: (JD) H. H. Torres Martínez (Posgrado en Ciencias Bioquímicas de la UNAM). Tesis de Maestría: Análisis del desarrollo de la raíz en la mutante sl de Arabidopsis thaliana. 29 de Enero de 2014. (JD) B. J. Reyes Hernández (Posgrado en Ciencias Biomédicas de la UNAM). Tesis de Doctorado: Mapeo genético y caracterización fenotípica de una mutante de Arabidopsis thaliana afectada en el desarrollo de la raíz. 15 de Octubre de 2014. Mención Honorifica. (SS) C.A. Sierra Sarabia (Licenciatura en Ciencias Biológicas, UAEM). Regeneración de raíces a partir de los callos de Ferocactus peninsulae, una cactácea desértica. 21 de agosto de 2015. (SS) J. Pacheco Mendoza (Licenciatura en Ciencias, UAEM). Obtención de callos y raíces transgénicas de las cactáceas desérticas Stenocereus gummosus y Ferocactus peninsulae. 4 de septiembre de 2015. (SS) M. L. López Valle (Posgrado en Ciencias Bioquímicas, UNAM). Tesis de Maestría: Participación de genes de la subfamilia PLETHORA en el crecimiento determinado de la raíz del cardón Pachycereus pringlei. 8 de Octubre de 2015. Mención Honorífica. Donativos: (SS) 2012-2014, Proyecto financiado por DGAPA, UNAM (IN2049012): “Estudio de la regulación genética del crecimiento determinado de la raíz de cactáceas desérticas”. Monto del periodo: $250,000.00. 2013-2014, Proyecto financiado por SEP-CONACyT (128135): “Integration of nodulation signalling pathway and assessment of its performance in promoting nitrogen-fixing rhizobial symbiosis in rice”. Monto del periodo: $375,000.00. 2015-2017, Proyecto financiado por DGAPA, UNAM (IN207115): “Análisis del transcriptoma del cardón Pachycereus pringlei para elucidar los mecanismos genéticos que regulan el crecimiento determinado de la raíz de cactáceas desérticas”. Monto del periodo: $260,000.00. 2015-2018, Proyecto financiado por SEP-CONACyT (240055): “Exploración de los mecanismos de regulación del crecimiento determinado de la raíz de cactáceas desérticas”. Monto del periodo: $1,051,000.00. 2015-2017, Proyecto financiado por FOINS-CONACyT (219522): “Análisis de tipo de crecimiento de la raíz primaria en Cactaceae”. Monto del periodo: $115,000.00. (JD) 2011-2014, Proyecto financiado por SEP-CONACyT (127957): “Caracterización y mapeo genético de las mutantes de Arabidopsis thaliana afectadas en el desarrollo del sistema radical: Parte II”. Monto del periodo: $1,300,000.00 2012-2014, Proyecto financiado por DGAPA, UNAM (IN204312) “Análisis del desarrollo de la raíz de Arabidopsis thaliana a nivel genético y celular”. Monto del periodo: $392,720.00 2014-2015, Proyecto de Estancia Sabática en extranjero financiado por CONACyT y PASPADGAPA "Análisis celular de la iniciación y la morfogénesis de la raíz lateral en Arabidopsis thaliana". Apoyo de CONACyT, el monto: 24,000 USD. Apoyo de PASPA-DGAPA, el monto: $16,560 y el apoyo para boletos de avión. 2015-2017, Proyecto financiado por DGAPA, UNAM (IN205315) “Regulación genética y bases celulares de desarrollo del sistema radical en Arabidopsis thaliana”. Monto del periodo: $251,900.00 (primer año) 2015-2017, Proyecto financiado por SEP-CONACyT: “La morfogénesis del primordio de la raíz lateral en Arabidopsis thaliana: análisis celular y regulación genética” (No. 237430). período: $2,000,000.00 Participación en docencia: (SS) Participación en 3 cursos de Posgrado en ciencias BQ y 1 curso de Licenciatura, en Comités Tutorales de 9 estudiantes de Maestría y Doctorado y 1 de Licenciatura, miembro de jurado de 6 exámenes de estudiantes de Posgrado. (JD) Participación en 2 cursos de Posgrado en ciencias BQ, UNAM y en un Tópico Selecto de Posgrado en Ciencias Biológicas de la UNAM, en Comités Tutorales de 5 estudiantes de Maestría y Doctorado; participación como miembro de jurado en 3 exámenes de estudiantes de Posgrado). Divulgación: 1. Reyes Hernández B.J., Díaz de la Garza R.I., Dubrovsky J.G. 2015. Folatos: su síntesis, metabolismo, transporte y papel en el desarrollo de plantas. Revista de Educación Bioquímica, en prensa. 2. Entrevista con el Dr. J. Dubrovsky transmitida por TELEVISA, Programa: "Creadores Universitarios", Capítulo: por transmitirse en diciembre del 2015. El tema: "Plantas y Raíces", 4 de Noviembre de 2015. 3. Murphy-Perez F. Rodriguez-Alonso G. 2014. Fábrica de cristales: en el subsuelo, en los riñones y en el laboratorio. Hypatia, 49-50. Premios y reconocimientos: 1. Joseph Dubrovsky: Miembro de Sistema Nacional de Investigadores, nivel III. Otorgado por CONACyT. Mexcio. 01/01/2014 2. Svetalana Shiskova: Miembro de Sistema Nacional de Investigadores, nivel II. Otorgado por CONACyT. Mexcio. 01/01/2015 3. Premio AgroBio México a Blanca Jazmín Reyes Hernández, estudiante del Dr. J. Dubrovsky; Categoría: Mejor Tesis de Doctorado. Otorgado por AgroBio México. México. 01/01/2014 4. Mención honorifica para el trabajo de B.J. Reyes-Hernández, estudiante del Dr. J. Dubrovsky, que fue presentado en el Segundo Concurso de Fotografía Científica convocado por la Coordinación de la Investigación Científica y la Dirección General de Divulgación de la Ciencia, UNAM. 15/10/2014 Títulos y autores de los carteles que presentará el grupo: 1. Estudio mediante RNA-seq del agotamiento del meristemo apical de la raíz primaria de Pachycereus pringlei, un cactus del Desierto de Sonora. Rodríguez-Alonso G., Matvienko M., López-Valle M. L., Dubrovsky J. G., Shishkova S. Análisis del transcriptoma de la mutante atx1setm atx1-1 de Arabidopsis thaliana, afectada en el desarrollo de la raíz. Napsucialy-Mendivil, S., Shishkova, S., Dubrovsky, J.G. Grupo de Lorenzo Segovia (2014-2015) Título: Evolución y manipulación de la relación estructura-función de proteínas Resumen: Una de las metas de la biotecnología moderna es diseñar enzimas adecuadas para condiciones particulares. En el grupo seguimos varios enfoques para modular las propiedades y función de proteínas de interés. Estos enfoques permitentambién obtener información valiosa sobre las propiedades fisicoquímicas y estructurales de las proteínas. El número de secuencias ha crecido de una forma extremadamente rápida en años recientes lo cual permite, entre otras cosas, diseñar secuencias consenso las cuales se ha observado son más estables. Diseñamos un dominio Rossmann consenso el cual fusionamos al dominio catalítico silvestre de la Shikimato deshidrogenasa de E. coli (SDH), nuestra enzima modelo, obteniendo así una proteína consenso quimérica activa. La enzima purificada mostró tener constantes catalíticas esencialmente idénticas a la proteína silvestrey hemos observado por microcalorimetría que hay dos componentes aparentes en la curva que al ser desconvolucionados indican la presencia de un segundo componente de desnaturalización 10 grados mayor que en la silvestre el cual correspondería a la consenso. Por otro lado hemos utilizado una paquetería llamada Rosetta Enzyme Design aunado a un extenso análisis de secuencias y estructuras para cambiar la especificidad por el cofactor de la SDH de NADP a NAD. Hemos obtenido construcciones las cuales han perdido casi completamente la afinidad por NADP aunque tienen baja afinidad por NAD. Otros proyectos del laboratorio estudian la estructura y actividad de expansinas que son proteínas no hidrolíticas que actúan sobre los polisacáridos de la pared celular de las plantas. La superfamilia de expansinas incluye genes de organismos eucariotes (plantas, hongos y nemátodos) y bacterianos. Analizando la estructura de expansinas bacterianas encontramos que además de los dominios canónicos (D1 y D2) y los aminoácidos conservados que forman la superficie de unión al sustrato, también contienen residuos de superficie con carga eléctrica que permite clasificarlas en ácidas o básicas. El mapeo de la distribución de carga permitió identificar un patrón general en el que la zona donde reside la función de las expansinas (D1) es negativa mientras que el lado opuesto de la molécula es menos negativo o es positivo. Para conocer si existe una razón funcional de este diferencial electrostático estudiamos la expansina Exlx1 del hongo Schizophyllum commune que también tiene la característica positivo/negativo del D1, y cuya actividad es fácilmente medida a través de una celulasa reportera. La generación de variantes con mayor o menor diferencia de carga permitirá conocer si el nivel de actividad se modifica. Por otro lado, también analizamos la contribución de dominios proteicos extra a la unión al sustrato y la actividad de otra expansina: Exl1 de la bacteria Sorangium cellulosum, que a través de un linker no estructurado de 100 residuos se conecta a un módulo de unión a celulosa. Nuestro interés es caracterizar la unión de la proteína silvestre y la que carece del linker y el CBM, y el efecto de modificaciones postraduccionales en el linker sobre la actividad. Integrantes del Grupo: Lorenzo Segovia Claudia Martínez Ernesto Pérez Rueda (Hasta Julio de 2014) Mabel Rodríguez (Post-Doctoral) Blanca Ramos (Técnico desde sept. 2015) Miguel Olarte (Doctorado) Fernando García (Doctorado) Iris Bravo (Maestría) Karen Acosta (Maestría) Sofía Mendoza (Licenciatura) Luis Acosta (Licenciatura) Marcela Ayala (Hasta Nov de 2014) Mayra Avelar (Doctorado) Estefanía Sierra (Doctorado) Joaquín Ramírez (Doctorado) Publicaciones (Martínez y/o Segovia): 1. García-Guevara F., Avelar M., Ayala M., and Segovia L. 2015. Computational Tools Applied to Enzyme design- a review. Biocatalysis. Accepted. 2. Ayala M., Segovia L., and Torres E. 2015. Halogenases: A Biotechnological Alternative for the Synthesis of Halogenated Pharmaceuticals. Mini-Reviews in Medicinal Chemistry. In press. 3. Pérez-Landero S., Sandoval-Motta S., Martínez-Anaya C., Yang R., Folch-Mallol J., L.M., Ventura L., Guillén L., Aldana-González M., Nieto-Sotelo J. 2015. Complex regulation of Hsf1Skn7 activities by the catalytic subunits of PKA in Saccharomyces cerevisiae. BMC Systems Biology, 9:42. 4. Pastor N, Dávila S, Pérez-Rueda E., Segovia L., Martínez-Anaya C. 2015. Electrostatic analysis of bacterial expansins. Proteins: Struct Funct Bioinf, 83, 215-223. 5. Armenta-Medina D., Segovia L., and Perez-Rueda E. 2014. Comparative genomics of nucleotide metabolism: a tour to the past of the three cellular domains of life. BMC Genomics, 15:800 6. Olarte-Lozano M., Mendoza-Nuñez M.A., Pastor N., Segovia L., Folch-Mallol J, MartínezAnaya C. 2014. PcExl1 a novel acid expansin-like 1 protein from the plant pathogen Pectobacterium carotovorum, binds cell walls differently to BsEXLX1. PLOS One, 9, e95638 Alumnos Graduados: Iris Bravo (Maestría 2015) Mario A. Mendoza (Maestría 2015) Luis A. Acosta (Licenciatura 2015) Vanessa Gómez (Licenciatura 2014) Donativos: 1. CONACYT 132580 Análisis evolutivo y diseño de dominios Rossmann en la superfamilia de las Aminoácido Deshidrogenasas. L. Segovia 2. CONACYT 166050 Caracterización de la relación estructura-función de proteínas con actividad modificadora de la celulosa. C. Martínez 3. DGAPA IN213511 Uso de información evolutiva para diseñar quimeras de proteínas. L. Segovia 4. SENER-CONACYT 201441 Fondo de Hidrocarburos “Plataformas de observación oceanográfica, línea base, modelos de simulación y escenarios de la capacidad natural de respuesta ante derrames de gran escala en el Golfo de México” L. Pardo, Resp Institucional. Participación en docencia: Posgrado en Ciencias Bioquímicas, IBT. Curso de Biología Molecular. Curso de Estructura y Función de Proteínas. Curso de Biocatálisis. Curso de Biología Celular Curso “Del gen al producto” Licenciatura de Biología, Facultad de Ciencias UNAM Taller de “La Biología a partir de las biomoléculas, nuevos paradigmas y fronteras” Facultad de Farmacia UAEM Curso Escritura de Artículos Científicos Divulgación: 1. Martínez-Anaya C., López-Munguía A. La celulosa: fibra y energía. Academia de Ciencias de Morelos, A.C. La Unión de Morelos. 15 de junio 2015 2. Folch-Mallol J., Ayala-Aceves M., Martínez-Anaya C., Cossio-Bayugar R. Enfermedades vemos, virus ¿qué sabemos? Academia de Ciencias de Morelos, A.C. La Unión de Morelos. 18 de mayo 2015. 3. Martínez Anaya C. (2015) La bioinformática a la ayuda de la Bioquímica. Estudio teórico para la elucidación de la actividad de proteínas. Revista Biotecnología en Movimiento, IBT-UNAM No. 1 Abril-Junio, 2015. 4. Martínez-Anaya C., García-Guevara F. (2014). Ingeniería de Proteínas para el mejoramiento de enzimas. Revista Digital Universitaria Vol. 15 No.12. 5. Martínez-Anaya C. (2014). ¡Bacterias donde ni te imaginas! Contribución al suplemento semanal de la revista Recreo editada por Investigación y Desarrollo ID. Distinciones: 1. Miembro del Comité Organizador del 4th International Workshop: Frontiers in Protein Folding, Evolution and Function- 5º Congreso de la rama de Fisicoquímica, Estructura y Diseño de Proteínas de la Sociedad Mexicana de Bioquímica. (2015) 2. Vocal de la Mesa Directiva de la Academia de Ciencias de Morelos- Periodo 2013-2015 Títulos y autores de los cárteles que presentará el grupo: 1. Comparative Analyses of plant expansins: EXPA vs. EXPB. Perla Acosta-Maldonado, Nina Pastor, Lorenzo Segovia, Claudia Martinez Anaya. 2. Redesign of the Shikimate Dehydrogenase enzyme, invertion on substrate specificity. Fernando Garcia Guevara, Lorenzo Segovia Grupo de Lorival Possani (2014-2015) Investigador emérito: Dr. Lourival Domingos Possani Postay Título: Proteínas, péptidos y genes del veneno de alacranes y anticuerpos protectores. Resumen; En los últimos 41 años mi laboratorio ha trabajado de forma preponderante varios aspectos que se refieren a los componentes del veneno de alacranes. Existen varias razones para realizar este tipo de investigación. En primer lugar está el interés médico por el elevado número de accidentes que ocurren en el país debido al piquete de alacranes peligrosos a los humanos. Se estima que el número de casos registrados en los hospitales y centros médicos del país es del orden de trescientos mil accidentes anuales, datos que se pueden verificar en las estadísticas de la Dirección General Adjunta de Epidemiología de la Secretaria de Salud. En segundo lugar está el interés científico en estudiar animales con una larga historia evolutiva. Se estima que los alacranes aparecieron en la tierra hace más de 400 millones de años y han podido desarrollar mecanismos para sobrevivir y conquistar los más variados nichos ecológicos. Nos interesa saber que constituyentes moleculares (principalmente proteínas y ácidos nucléicos) les permitieran tal diversidad y sobrevivencia. Han desarrollado mecanismos moleculares que les permiten a atrapar sus presas, de las cuales se alimentan y defenderse de posibles predadores. En el desarrollo de este trabajo descubrimos que entre los componentes de sus venenos están péptidos que selectivamente afectan la comunicación celular, causando el bloqueo o la modificación de las cinéticas de apertura y cierre de poros formados por proteínas de membrana conocidas como “canales iónicos”, que son fundamentales en la comunicación entre células de los organismos pluricelulares. Encontramos péptidos con actividades antibióticas, antiparasitarias, antiepilépticas, analgésicas y moduladoras de muchas funciones biológicas, que los hacen instrumentos importantes para estudios de fenómenos neurológicos, musculares e inmunológicos. De misma forma el conocimiento de los componentes moleculares del veneno de los alacranes nos ha permitido diseñar estrategias para la generación de anti-venenos. El conocimiento de la estructura y función de los componentes del veneno abrió la posibilidad de visualizar su posible uso como drogas o fármacos que pueden llegar a ser útiles tanto en términos de la salud humana, como animal y vegetal. Cabe mencionar que algunos componentes no son tóxicos para mamíferos, pero son eficientes en el control de otros artrópodos que pueden ser plagas agrícolas o vectores de enfermedades como paludismo y ciertas enfermedades causadas por virus. De esta forma el estudio bioquímico de estos venenos y la obtención del proteoma y transcriptoma completos de varias especies de alacranes han servido como modelos para la incursión en estudios de otras áreas del conocimiento. Los datos obtenidos durante los años 2014 y 2015 fueron reportados en 21 artículos científicos publicados en revistas internacionales indizadas, tres capítulos de libro, se publicó como coeditor un libro por la Editorial Alemana Springer: “Venenos de alacranes”. Se concedieron 6 patentes y se depositaron 3 nuevas patentes. Las tesis terminadas fueron 5 (2 Doc, 2 Ms, 1 Lic). Integrantes del grupo 2014-1015 Investigadores Asociados Dra. Georgina Gurrola Briones 1" " Posdoctorales Dra. Rita Restano-Cassulini Dra. Verónica Quintero Hernández Dra. Maria Martha Pedraza Escalona Dra. Juana Maria Jiménez Vargas Dr. David Jauregui Zuñiga Dra. Lidia González Morales Dr. Carlos E. Santibáñez López Técnicos Académicos Dr. Fernando Zamudio Zuñiga Téc. Fredy I. Coronas Valderrama Estudiantes de Licenciatura Ivett Uribe Mendoza Estudiantes de Posgrado Dra. Itzel Amaro Estrada M. en C. José Ignacio Veytia Bucheli QBP Jimena Isaias Cid Uribe Biól. Rodolfo Rodríguez Ravelo Biól. Rosby del Carmen Najara Meza M. en C. Leonel Vargas Jaimes M. en C. Maria Teresa Romero Gutiérrez M. en C. Karen Sofia Luna Ramírez Personal Administrativo Biól. Cipriano Balderas Altamirano Linda Solaris Espinosa C.P. Carmen Martínez Segura Publicaciones: 1. Rodríguez-Ravelo, R.,Restano-Cassulini, R., Zamudio F.Z., Coronas,F.I.V., Espinosa-López, G., Possani, L.D. A K+ channel blocking peptide from the Cuban scorpion Rhopalurus garridoi. Peptides 53:42-47 (2014). 2. Luna-Ramírez, K., Sani, M.A., Silva-Sánchez, J., Jiménez-Varga, J.M., Reyna-Flores, F., Winkel, K.D., Wright. C.E., Possani, L.D., Separovic, F. Membrane interactions and biological activity of antimicrobial peptides from Australian scorpion. Biochim. Biophys. Acta 1838:21402148 (2014). 3. Ortiz, E., Rendón-Anaya, M., Rego, S.C., Schwartz, E.F., Possani, L.D. Antarease-like Znmetalloproteases are ubiquitous in the venom of different scorpion genera. BBA – General Subjects 1840:1738-1746 (2014). 2" " 4. Vergara, I., Pedraza-Escalona, M., Paniagua, D., Restano-Cassulini, R., Zamudio, F., Batista, C.V.F., Possani, L.D., Alagon, A. Eastern coral snake Micrurus fulvius venom toxicity in mice is mainly determined by neurotoxic phospholipase A2. J. Proteomics 105:295-306 (2014). 5. Luna-Ramirez, K., Bartok, A., Restano-Cassulini, R., Quintero-Hernández, V., Coronas, F.I.V., Christensen, J., Wright, C.E., Panyi, G., Possani, L.D. Structure, molecular modeling and function of a novel potassium channel blocker, urotoxin, isolated from the venom of the Australian scorpion Urodacus yaschenkoi. Molecular Pharmacology 86:28-41 (2014). 6. Pedraza-Escalona, M., Batista, C.V.F., Restano-Cassulini, R., Sandoval-Rios M., Coronas, F.I.V, Possani, L.D. A proteomic analysis of the early secondary molecular effects caused by Cn2 scorpion toxin on neuroblastoma cells. J. Proteomics 111: 212-223 (2014). 7. González-Morales, L., Pedraza-Escalona, M., Diego-García, E., Restano-Cassulini, R., Batista, C.V.F., Gutierrez, M.C., Possani, L.D. Proteomic characterization of the venom and transcriptomic analysis of the venomous gland from the Mexican centipede Scolopendra viridis. J. Proteomics 111:224-237 (2014). 8. Picco, G., Corzo, G., Possani, L.D., Prestipino, G. Interaction of the scorpion toxin Discrepin with Kv4.3 channels and A-type K+ channels in cerebellum granular cells. BBActa – General Subjects 1840:2744-2751 (2014). 9. Coronas, F.I.V., Diego-Garcia, E., Restano-Cassulini, R., de Roodt, A.R., Possani, L.D. Biochemical and physiological characterization of a new Na+-channel specific peptide from the venom of the Argentinean scorpion Tityus trivittatus. Peptides, 68:11-16 (2014) http://dx.doi.org/10.1016/ j.peptides.2014.05.002 (2014). 10. Ramírez-Carreto,S. Jimenez-Vargas,J.M. Rivas-Santiago,B. Corzo,G. Possani,L.D. Becerril,B. Ortiz,E. 2015. Peptides from the scorpion Vaejovis punctatus with broad antimicrobial activity Peptides, 73, 51-59. 11. Santibáñez-Lopez,C.E. Possani,L.D. 2015. Overview of the Knottin scorpion toxin-like peptides in scorpion venoms: Insights on their classification and evolution Toxicon, Jul 14 [Epub ahead of print], . 12. Rodríguez-Ravelo,R. Batista,C.V. Coronas,F.I. Zamudio,F.Z. Hernández-Orihuela,L. Espinosa-Lopez,G. Ruiz-Urquiola,A. Possani,L.D. 2015. Comparative proteomic analysis of male and female venoms from the Cuban scorpion Rhopalurus junceus Toxicon, Jul 10 [Epub ahead of print] . 13. Ortiz,E., Possani,L.D. 2015. The unfulfilled promises of scorpion insectotoxins Journal of Venomous Animals and Toxins Including Tropical Diseases, 21, . 14. Luna-Ramirez,K. Quintero-Hernandez,V. Juarez-Gonzalez,V.R. Possani,L.D. 2015. Whole Transcriptome of the Venom Gland from Urodacus yaschenkoi Scorpion PLoS ONE, 10, e0127883. 15. Espino-Solis,G.P. Quintero-Hernandez,V. Olvera-Rodriguez,A. Calderon-Amador,J. Pedraza-Escalona,M. Licea-Navarro,A. Flores-Romo,L. Possani,L.D. 2015. Novel monoclonal antibody against alphaX subunit from horse CD11c/CD18 integrin Veterinary Immunology and Immunopathology, 164, 220-226. 16. Quintero-Hernandez,V. Ramirez-Carreto,S. Romero-Gutierrez,M.T. Valdez-Velazquez,L.L. Becerril,B. Possani,L.D. Ortiz,E. 2015. Transcriptome Analysis of Scorpion Species Belonging to the Vaejovis Genus PLoS ONE, 10, e0117188. 3" " 17. Okada,M. Corzo,G. Romero-Perez,G.A. Coronas,F. Matsuda,H. Possani,L.D. 2015. A pore forming peptide from spider Lachesana sp. venom induced neuronal depolarization and pain Biochimica et Biophysica Acta (BBA), 1850, 657-666. 18. Ortiz,E. Gurrola,G.B. Schwartz,E.F. Possani,L.D. 2015. Scorpion venom components as potential candidates for drug development Toxicon, 93, 125-135. 19. Coronas,F.I. Diego-Garcia,E. Restano-Cassulini,R. de Roodt,A.R. Possani,L.D. 2015. Biochemical and physiological characterization of a new Na-channel specific peptide from the venom of the Argentinean scorpion Tityus trivittatus Peptides, 68, 11-16. 20. Riaño-Umbarila, L., Ledezma-Candanoza, L.M., Serrano-Posada, H., Fernández-Taboada, G., Olamendi-Portugal, T., Rojas-Trejo, S., Gómez-Ramírez, I., Rudiño-Piñera, E., Possani, L.D., Becerril, B. Optimal neutralization of Centruroides noxius venom is understood through a structural complex between two antibody fragments and the Cn2 toxin. J. Biol. Chem. Disponible in linea: http://www.jbc.org/cgi/doi/10.1074/jbc.M115.685297 (2015). 21. Zhao, Y.T., Valdivia, C.R., Gurrola, G, Powers, P.P., Willis, B.C., Moss, R.L. Jalife, J., Valdivia, H.H. Arrhythmogenesis in a catecholaminergic polymorphic ventricular tachycardia mutation that depresses ryanodine receptor function. Proceedings of the National Academy of Sciences of USA, 112 No., E1669 (2015. 22. Zhao, Y.T. Valdivia, C.R., Gurrola, G., Hernández, J.J., Valdivia H.H. Arrhytmogenic mechanisms in ryanodine receptor channelopathies. Science China Life Sciences, 58, No.,54 (2015). Edición de libro Handbooks of Toxinology, Editor-in Chief P. Gopalakhrisnakone, Editorial Springer (Alemania). El libro “Scorpion venoms”, editores: L.D.Possani, E.F. Schwartz, R.Rodríguez de la Vega: 23 capítulos con 115 figuras, 38 tablas, con un total: 575 páginas (printed format: 2015). Capítulos de libro 1. Abdel-Rahman,M.A. Quintero-Hernandez,V. Possani,L.D. 2014. Scorpion Venom Gland Transcriptomics and Proteomics: An Overview en: Calvete,J.J. Venom Genomics and Proteomics. Springer Netherlands. pags. 1-17. 2. Rodriguez Rodriguez, E.R., Riaño Umbarila, L.,Possani,L.D. y Baltazar Becerril. Anti-venoms: Recombinant neutralizing antibodies, a new generation of scorpion anti-venoms. Book Scorpion venom. Handbook of Toxinology, Springer, chapter 6, pp.139-159, (2015). 3. Rodríguez de la Vega, R.C., Corzo, G., Possani, L.D. Scorpion venoms as a platform for drug development. G.F. King editor, libro: Venom to Drugs, RSC Drug Discovery, Royal Society of Chemistry, chapter 7, pages 204-220 (2015). Alumnos graduados Licenciatura: 1. Para la obtención del título de Bióloga de la Facultad de Ciencias Biológicas de la Universidad Autónoma del Estado de Morelos: “Estudio de la interacción de la Imperatoxina A con el receptor de la Ryanodina por medio de ensayos de entrecruzamiento” por Ivett Uribe Mendonza. El tutor principal fue la Dra. Georgina Gurrola Briones. Fecha: 6 de noviembre de 2015. Maestria: 4" " 2. Para la obtención del grado de Maestro en Ciencias Bioquímicas del Instituto de Biotecnología – UNAM “Efecto inmunomodulador de la toxina Vm24 del alacrán Vaejovis mexicanus el linfocitos T de memoria efectores CD+”, por el Q.F.B. José Ignacio Veytia Bucheli. Fecha: 31 de Julio de 2015. 3. Para la obtención del grado de Maestro en Ciencias del Instituto de Investigación en Ciencias Basicas y Aplicadas, Centro de Investigación en Dinámica Celular de la Universidad Autónoma del Estado de Morelos, bajo el título: “Expresión heteróloga de la toxina de calcio intrepicalcina, proveniente del veneno del alacrán Vaejovis intrepidus y caracterización de su actividad biológica”, presentado por Leonel Vargas Jaimes, bajo la tutoria de la Dra. Verónica Quintero Hernández. Fecha: 16 de octubre de 2015. Doctorado: 4. Para la obtención del grado de “Doctor of Philosophy” del Departamento de Farmacología y Terapeutica, Facultad de Medicina, Odontologia y Ciencias de la Salud, de la Universidad de Melbourne, Australia, con la tesis: “Urodacus yaschenkoi, Autralian scorpion: phylogeny, venom characterization and pharmacology”. Fungí como co-tutor de la alumna Karen Sofia Luna Ramirez. Fecha febrero de 2014. 5. Para la obtención del grado de Doctor en Ciencias (Instituto de Biotecnología, Universidad Nacional Autónoma de México): “Obtención de anticuerpos de origen humano que neutralicen la toxina Ts1 del alacrán brasileño Tityus serrulatus” por la M. en C. Itzel Amaro Estrada.Fecha de presentación: 5 de febrero de 2015. Patentes concedidas Fases Nacionales concedidas de la Solicitud Internacional WO2008139243 A1, No. inicial: PCT/IB2007/001544: “Vm23 Y Vm24, dos péptidos del veneno de alacrán que bloquean con alta selectividad a los canales de potasio (subtipo Kv1.3) de los linfocitos T humanos y disminuyen la reacción in vivo de DTH en ratas”. L. D. Possani P., G. Gurrola-Briones, S. P. Salas-Castillo, C. V. Ferreira B., Z. S. Varga, G. Panyi, R. Gáspár. 1. 2007353147B2 Concedida en Australia el 17 de julio de 2014. Solicitud Internacional WO2008139243 A1. 2. EA020131B1 Concedida en Euroasia el 06 de junio de 2014. Solicitud Internacional WO2008139243 A1. 3. CN101796069B Concedida en China el 19 de febrero de 2014. Solicitud Internacional WO2008139243 A1. 4. No. 10-1524517 concedida en Corea el 26 de mayo de 2015. Solicitud Internacional WO2008139243 A1. 5. MX 325627 “Familia de variantes de anticuerpos recombinantes humanos que neutralizan a las toxinas de alacrán CN2 Y CSS2 así como a los venenos respectivos: Centruroides Noxius y Centruroides suffusus suffusus”. Lidia Riaño U., Everardo R. Rodríguez R., Baltazar Becerril L.y Lourival D. Possani P. Concedida en México el 13 de noviembre de 2014. 6. MX320050B “Nuevo péptido antibiótico híbrido y sus variantes”. Georgina Gurrola B., Lorenzo Sánchez V., Jesús Silva S. y Lourival D. Possani P. Concedida en México el 12 de mayo de 2014. Patentes depositadas en México (IMPI) 5" " 1. PCT/MX2014/000093. “Nuevos anticuerpos monoclonales contra el receptor DEC-205 de células dendríticas de pollo”. Possani P. L. D., Pedraza E. M. Martha, Espino S. P. G., Olvera R. A. y Cardoso T. H. M. Solicitud internacional presentada el 17 de junio de 2014. 2. MX/a/2015/008582. “Péptidos del alacrán Vaejovis punctatus como antibióticos contra Mycobacterium tuberculosis y otras bacterias y levaduras patógenas”. Ernesto Ortiz, Santos Ramírez, Juana M. Jiménez, Gerardo A. Corzo, Baltazar Becerril y Lourival D. Possani, responsable Dr. Possani. Solicitud presentada el 1 de Julio de 2015. 3. MX/a/2015/011378. “Familia de variantes de anticuerpos recombinantes humanos que neutralizan a los principales venenos mexicanos”. Inventores: Lidia Riaño Umbarila, Everardo Remi Rodríguez Rodríguez, Ilse Viridiana Gómez Ramírez, Jonathan Noé Arredondo López, Timoteo Celso Olamendi Portugal, Guillermo Fernández Taboada, Lourival Domingos Possani Postay, Baltazar Becerril Luján, responsable Dr. Becerril. Solicitud presentada el 2 de Septiembre de 2015. Participación en docencia 1. Colaboración como maestro del Curso Básico de Bioquímica a nuestro Postgrado. 2. Tutor de varios alumnos de nuestros posgrados y de otras Universidades. Divulgación En 2014: Eventos : VI Simposio Cinvestav/Sigma-Aldrich Difusión : Nacional Tipo de participación : Ponente Tipo de presentación : oral Lugar : Cinvestav-IPN Fecha de Inicio : 28/mayo/2014 Fecha de Término : 29/mayo/2014 Título : Ponencia plenaria titulada: "Aspectos bioquímicos del veneno de alacranes:estructura y función" Autor 1 : [L.D.Possani ] Dr. Lourival Domingos Possani Postay Tipo de Actividad : Feria Eventos : Expo-DIC Diclab 2014 Difusión : Nacional Tipo de participación : Ponente Tipo de presentación : oral Lugar : Centro Internacional de Exposiciones y Convenciones Fecha de Inicio : 18/septiembre/2014 Fecha de Término : 19/septiembre/2014 Título : Conferencia plenaria, titulada. "Avances recientes en el conocimiento de la estructura y función de componentes del veneno de alacranes" Autor 1 : [L.D.Possani ] Dr. Lourival Domingos Possani Postay 6" " Tipo de Actividad : Simposio Eventos : Primer simposio internacional de neurotoxicidad y neuroprotección Difusión : Internacional Tipo de participación : Ponente Tipo de presentación : oral Lugar : Raquet Club - Cuernavaca Fecha de Inicio : 25/junio/2014 Fecha de Término : 27/junio/2014 Título : Conferencia plenaria titulada: "Avances cientificos en el conocimiento de la estructura y función de componentes del veneno de alacranes" Autor 1 : [L.D.Possani ] Dr. Lourival Domingos Possani Postay Tipo de Actividad : Curso Eventos : Primer curso de animales venenosos en México: Biología y Clínica Difusión : Nacional Tipo de participación : Ponente Tipo de presentación : oral Lugar : Instituto de Biotecnología - UNAM, Cuernavaca Fecha de Inicio : 18/septiembre/2014 Fecha de Término : 20/septiembre/2014 Título : Ponencia de una hora sobre: "Aspectos bioquímicos del veneno de alacranes:estructura y función" Autor 1 : [L.D.Possani ] Dr. Lourival Domingos Possani Postay Tipo de Actividad : Simposio Eventos : Simposio Nacional de Animales Venenosos Difusión : Nacional Tipo de participación : Ponente Tipo de presentación : oral Lugar : Fac. de Ciencias de la UNAM - Mexico-D.F. Fecha de Inicio : 25/noviembre/2014 Fecha de Término : 25/noviembre/2014 Título : Ponencia plenaria titulada: "Aspectos bioquímicos del veneno de alacranes:estructura y función" Autor 1 : [L.D.Possani ] Dr. Lourival Domingos Possani Postay En 2015: Ponencia: Caracterización de los componentes del veneno de alacranes. Presenté la conferencia plenaria de este simposio internacional.. Participación oral como Ponente. Participantes L.D.Possani . Sede Puebla, (16/06/2015-17/06/2015). Ponencia: Caracterización de los componentes del veneno de alacranes. Participación oral como Ponente. Participantes L.D.Possani . Sede Fac. de Químcas . UNAM, (04/09/2015-04/09/2015). 7" " Ponencia: Electrophysiological analysis of anti-venom efficacy against North African scorpions. Poster presentado en el 18th World Congress of the International Society on Toxinology. Participación cartel como Co-autor. Participantes R. Restano , Jorge, Walter,L.D.Possani . Sede Oxford, Inglaterra, (25/09/2015-30/09/2015). Ponencia: Transcriptome analysis of venomous glands of male and female scorpions of the species Centruroides limpidus. Poster 28-4-3 (página 114) presentado en el 18th World Congress of the International Society on Toxinology.Participación cartel como Co-autor. Participantes J.Cid ,E.Ortiz-Suri ,M.Romero ,L.D.Possani . Sede Oxford, Inglaterra, (25/01/2015-30/01/2015). Ponencia: Insights on the transcriptomic analysis of the venomous gland of the scorpion Thorellius atrox. Poster 28-4-9 (página 118) presentado en el 18th World Congress of the International Society on Toxinology. Participación cartel como Co-autor. Participantes M.Romero, V.Quintero, F.Sanchez-Flores, E.Ortiz-Suri, L.D.Possani. Sede Oxford, Inglaterra, (25/09/2015-30/09/2015). Donativos: 1. Proyecto IN200113 - DGAPA-UNAM (MN$ 260000 cada año 2014 y 1015)- PAPPIT con Titulo: “Aislamiento y caracterización de componentes del veneno de alacranes”. Último año de tres en 2015. 2. PEI-CONACyT 221835 (MN$ 1000000), donados por Instituto Bioclón S.A. de C.V. Título: Desarrollo y evaluación de un antiveneno para el tratamiento de picadura de alacrán de la región del Norte de África y Medio Oriente. Aprobado para únicamente para el año 2015. 3. PROYECTO P607: LABORATORIOS SILANES (MN$2000000 año 2015) Pago final del Convenio DGAJ-DPI-280214-824, que se refiere a la cesión de derechos de patente sobre la expresión heteróloga de toxinas recombinantes del veneno de alacranes para la generación de anti-venenos en caballo. 4. SEP-CONACyT 237864 (MN$ 1800000 total, pero MN$ 345000 en el año 2015). Titulo: Proteomica y transcriptomica de las glándulas del veneno de alacranes. Títulos y autores de los carteles que presentará el grupo: 1. Insights on the transcriptomic analysis of the venomous gland of the scorpion Thorellius atrox. María T. Romero-Gutiérrez1, Verónica Quintero-Hernández1, Alejandro SanchezFlores2, Ernesto Ortiz1, Lourival D Possani1 2. Transcritome analysis of venomous glands of male and female scorpions of the species Centruroides limpidus. Jimena Cid Uribe, Ernesto Ortiz, Maria T. Romero-Gutérrez, Lourival D. Possani. 3. Efecto inmunomodulador de la toxina Vm24 del alacrán Vaejovis mexicanus en linfocitos T de memoria efectores CD4+. JI. Veytia-Bucheli, JM. Jiménez-Vargas, EI. Melchy-Pérez, R. Restano-Cassulini, GB. Gurrola, Y. Rosenstein, LD. Possani. 8" " Grupo de Luis Covarrubias (2014-2015) Título: El recambio en la esencia de los mecanismos de envejecimiento y longevidad. Resumen: Diversos procesos durante la embriogénesis determinan cualitativa- y cuantitativamente la composición del organismo adulto. Así entonces, por ejemplo, pequeñas deficiencias en el proceso de diferenciación celular causado por una combinación de alteraciones genéticas o epigenéticas pueden limitar la calidad de vida. Por otro lado, la calidad de vida desde el nacimiento también depende del buen mantenimiento de sus componentes tanto moleculares como celulares. Nuestra propuesta es que la capacidad de recambio tanto molecular como celular se encuentra en el centro de los mecanismos que controlan la longevidad y el envejecimiento, y contribuye al desarrollo de las enfermedades asociadas. El estrés oxidativo y el metabolismo. Hemos estudiado las especies reactivas de oxígeno como reguladoras de la muerte celular, pero considerando también sus efectos en la senescencia replicativa. Como consecuencia de esta área de investigación, descubrimos el efecto notable de las especies reactivas de oxígeno en el metabolismo. En particular, identificamos al peróxido de hidrógeno (H2O2) en el hígado como un regulador que previene de las alteraciones metabólicas que surgen durante la obesidad. Interesantemente, un régimen de ayuno o restricción de alimento a recién nacidos reduce marcadamente sus expectativas de vida indicando que los cambios metabólicos observados ponen en riesgo la vida del organismo. Relevante en el contexto del envejecimiento y la longevidad, los ratones carentes de catalasa no muestran un incremento marcado en el daño oxidativo y, paradójicamente, previene del daño oxidativo observado en el hígado bajo consumo de una dieta alta en grasas. Además, estos ratones mueren prematuramente alcanzando una vida máxima de 24 meses en comparación con los cerca de 30 meses de vida de animales silvestres. Esta muerte prematura no está asociada a indicadores de envejecimiento o a alguna enfermedad particular evidente (e.g., cáncer), lo que pudiera representar una separación entre los mecanismos que regulan la longevidad y los que causan el envejecimiento. Los oncogenes y la renovación de los tejidos. Una de las características típicas de animales viejos es la disminución en la capacidad de renovación de sus tejidos y la de regeneración, ya de por sí limitada en mamíferos. En contraste, la frecuencia de formación de tumores incrementa con la edad siendo el cáncer una de las causas más importante de morbilidad y mortalidad de individuos viejos. Hemos generado ratones transgénicos que expresan los oncogenes E6/E7 del papilomavirus humano HPV16 con el fin inmortalizar células precursoras (i.e., inhibir la senescencia replicativa) y generar modelos animales del cáncer. Como consecuencia de estos estudios descubrimos que la expresión de estos oncogenes incrementa las capacidades regenerativas y posiblemente mantienen la renovación de algunos tejidos sin detectarse un cambio significativo en la frecuencia de formación de tumores. Hemos detectado a Sonic Hedgehog y al estradiol como importantes reguladores de la renovación y regeneración de los tejidos. Determinar las condiciones óptimas de renovación celular sin alteraciones tisulares puede ayudar a entender los factores que promueven el cáncer y limitan la longevidad. En el centro de estos estudios está el mantenimiento y la expansión de células troncales, proceso que puede involucrar la reprogramación epigenética o la rejuvenación del epigenoma. Integrantes del Grupo: Investigadores Luis Covarrubias Celina García Meléndrez Concepción Valencia García Anabel Maciel (posdoctorado) Alvaro Marín (visitante) Estudiantes Gilda Guerrero Flores (Doctorado en Ciencias Bioquímicas) José Raúl Pérez Estrada (Doctorado en Ciencias Bioquímicas) Dulce María Arzate Vázquez (Doctorado en Ciencias Biomédicas) Niurka Trujillo Paredes (Doctorado en Ciencias Bioquímicas) Verónica Rojo León (Doctorado en Ciencias Biológicas) Sergio Eliezer López Hernández (Maestría en Ciencias Bioquímicas) José Angel Martínez Sarmiento (Maestría en Ciencias Bioquímicas) Alberto Valeriano Guerrero (Maestría en Ciencias Bioquímicas) Ana Karen Mojica Ávila (Maestría en Ciencias Bioquímicas) Francisco Leyva Castro (Licenciatura, Facultad de Ciencias, UAEM) Karen Ivette de León Barrera (Licenciatura, Facultad de Ciencias, UNAM) Gladys Mondragón F. (Licenciatura, Facultad de Biología, UAEM) Jessica Danielly Medina (Licenciatura, Ciencias Genómicas, UNAM) Mayra Ruíz (Licenciatura, Ciencias Genómicas, UNAM) Colaboradores René Drucker-Colín Magda Guerra C (IFC-UNAM) Omar Collazo (IFC-UNAM) Susana Castro Obregón (IFC-UNAM) Andras Nagy (SLRI-MSH, Toronto Canada) David Hernández García (LPRT-IBT-UNAM) Chris Wood (LNMA-IBT-UNAM) Publicaciones: 1. Covarrubias L. 2014. De la Clonación Molecular a la Clonación de Animales (Reprogración Genómica para la Terapia Celular)”. En: Grandes retos del siglo XXI (Héctor Vasconcelos, Coordinador). Coordinación de Difusión Cultural, Dirección General de Publicaciones y Fomento Editorial, UNAM (ISBN: 978-607-02-5132-0). 99-105. 2. Niurka Trujillo-Paredes, Concepción Valencia, Gilda Guerrero-Flores, Dulce-María Arzate, José-Manuel Baizabal, Magda Guerra-Crespo, Ayari Fuentes-Hernández, Iván Zea-Armenta and Luis Covarrubias. 2015. Regulation of differentiation flux by Notch signalling determines the number of dopaminergic neurons in the adult brain. Biology Open (The Company of Biologist). En revision. 3. José Raúl Pérez Estrada, David Hernández-García, Susana Castro-Obregón, Luis Covarrubias. 2015. Reduction of lipogenesis by hydrogen peroxide prevents from obesity-associated steatosis and increases starvation-induced mortality. En preparación 4. Celina García-Meléndrez, David Hernández-García, Verónica Rojo, José-Raúl Pérez-Estrada, Concepción Valencia, Luis Covarrubias. Mouse ear regeneration as a model of cervical epithelium growth in response to E6E7 oncogenes and estradiol. En preparación. Donativos: 1. Consejo Nacional de Ciencia y Tecnología, Fondo SEP-Ciencia Básica (131031) “Las células troncales y la reprogramación genómica en el contexto de la diferenciación dopaminérgica y la enfermedad de Parkinson”. 2010-2016. 2. Programa de Apoyo a Proyectos De Investigacion e Innovación Tecnologica, Dirección General de Asuntos del Personal Académico, UNAM (IN209813): “Ratones transgénicos modelo de enfermedades prevalecientes en México”. 3. Programa de Apoyo a Proyectos De Investigacion e Innovación Tecnologica, Dirección General de Asuntos del Personal Académico, UNAM (IA200915) 4. Evaluación de la regeneración y la carcinogénesis en tiempo real. 2015-2018. 5. Consejo Nacional de Ciencia y Tecnología, Fondo SEP-Ciencia Básica (239891) “La angiogénesis en modelos murinos de regeneración”. 2015-2018. 6. Convenio de colaboración con Landsteiner Scientific. 2015Premios: Premio (1er lugar) CANIFARMA 2014 en Investigación Básica. Premio de Investigación Médica Dr. Jorge Rosenkranz 2014 en el Area Básica. Participación en docencia: La Biología del Desarrollo en la Era Genómica. Curso básico optativo para la licenciatura en biología de la Facultad de Ciencias. Impartido por LC. La Biología a partir de las biomoléculas: nuevos paradigmas y aplicacions. Facultad de Ciencias. Participantes LC y CG-M. Curso básico de Biología Celular (sección muerte células y biología del desarrollo). Participante LC. Divulgación: Covarrubias, L. Regenerative medicine for the brain: between the “can do” and the “may do” (Medicina regenerativa para el cerebro: entre lo realizable y lo disponible). Taller: Aspectos Éticosy Legales de la Investigacióncon Células Troncales y la Neuroética. México, D.F. Enero 13-15, 2014. Impartida por LC. Covarrubias, L. Reprogramación metabólica y epigenética: delas células troncales a las enfermedades. “Primer Día de Puertas Abiertas del IBT”. Cuernavaca, Morelos. Mayo 23, 2014. Impartida por LC. Covarrubias, L. Modelos animales y celulares para estudiar enfermedades humanas. Instituto Nacional de Medicina Genómica. México, D.F. Marzo 27, 2015. Impartida por LC. Conferencia Interna en el IBT para alumnos de la Universidad Autónoma del Estado de México. Noviembre de 2015. Impartida por CG-M. Títulos y autores de carteles que presentará el grupo: 1. Los oncogenes E6/E7 y el estradio promueven la regeneración de la oreja pro diferentes mecanismos. Celina García, Concepción Valencia, Mariana Werner, José Raúl Pérez y Luis Covarrubias. 2. Construcción de un vector policistrónico autoinducible para promover reprogramación celular. José Angel Martínez Sarmiento y Luis Covarrubias. 3. Evaluación de la neurogénesis inducida por células de cuerpos embrioides en la sustancia nigra de ratones adultos. Dulce-María Arzate-Vázquez, Magdalena Guerra-Crespo y Luis Covarrubias. Grupo de Mario Soberón (2014-2015) Título: La oligomerización de la toxina Cry1Ac esta ligada a diferentes mecanismos de resistencia en dos especies de insectos plaga. Resumen: Las toxinas Cry de Bacillus thuringiensis ejercen su actividad toxica a través de la formación de poros líticos en la superficie de las células intestinales. En nuestro grupo de investigación hemos propuesto un modelo del modo de acción que involucra la activación proteolítica de la protoxina de Cry1Ab a una forma activa con una estructura de tres dominios la cual interacciona en primer término con proteínas membranales ancladas por glicosil-fosfatidilinositol (GPI) como fosfatasa alcalina y aminopeptidasa. Proponemos que esta interacción aproxima a la toxina activa a la membrana de las células intestinales donde interacciona posteriormente con una proteína tipo caderina la cual facilita la eliminación de la hélice alpha-1 del amino terminal y la oligomerización de la toxina. El oligomero o pre-poro gana afinidad por las moléculas ancladas por GPI que facilitan su inserción a la membrana y la formación del poro lítico. Recientemente se ha descrito en 5 diferentes especies de insectos lepidopteros que resistencia a la toxina Cry1Ac esta ligada a mutaciones en un transportador tipo ABC (ABCC2) implicando su participación en el modo de acción de la toxina Cry1Ac. Sin embargo, su papel en el modo de acción no esta aun definido. En esta ocasión vamos a presentar el análisis comparativo de la oligomerización de la toxina Cry1Ac en membranas de la vellosidad apical purificada de poblaciones sensibles y resistentes a la toxina Cry1Ac de Pectinophora gossypiella y Plutella xylostella, las cuales están ligadas a mutaciones en el receptor caderina y en el transportador ABCC2, respectivamente. Nuestros datos muestran que tanto el transportador ABCC2 como la caderina estan involucrados en la oligomerización de la toxina Cry1Ac. Integrantes del Grupo: Isabel Gómez Gómez, Investigador Titular B Janette Onofre, Investigador Posdoctoral Gretel Flores Ramírez, Investigador Posdoctoral (Hasta Junio 2015) Blanca Inés García Gómez, Técnico Académico Titular C Lisbeth Cabrera, Técnico Académico (Por proyecto) Fernando Zúñiga Navarrete, Estudiante Doctorado (tutor MS) José Ocelotl Oviedo, Estudiante Doctorado (tutor MS) Arlen Peña-Cardeña, Estudiante Doctorado (tutor IG) Diana Laura Martínez de Castro, Estudiante Doctorado (tutor MS) Daniela Carmona León, Estudiante Maestría (tutor IG) Alan I. Jiménez Reyes, Estudiante Maestría (tutor MS) Francisca Villanueva, Estudiante Maestría (tutor MS) Publicaciones: Revistas indexadas 1. Cantón, P. E., López-Díaz, J. A., Gill, S. S., Bravo, A., Soberón, M. 2014. Membrane binding and oligomer membrane insertion are necessary but insufficient for Bacillus thuringiensis Cyt1Aa toxicity. Peptides. 53: 286-291 2. Portugal, L., Gringonten, J. L., Caputo, G. F., Soberón, M., Muñoz-Garay, C., Bravo, A. 2014 Toxicity and mode of action of insecticidal Cry1A proteins from Bacillus thuringiensis in an insect cell line, CF-1. Peptides. 53: 292-299 3. Gómez, I., Sanchez, J., Muñoz-Garay, C., Matus, V., Gill, S. S., Soberón, M., Bravo, A. 2014. Bacillus thuringiensis Cry1A toxins are versatile-proteins with multiple modes of action: Two distinct pre-pores are involved in toxicity. Biochemical Journal. 459: 383–396. 4. Monnerat, R., Pereira, E., Teles, B., Martins, E., Praca, L., Quiroz, P., Soberón, M., Bravo, A., Ramos, F., Soares, C. M. 2014. Synergistic activity of Bacillus thuringiensis toxins aginst Simulium spp larvae. Journal of Invertebrate Pathology. 121: 70-73. 5. Chavez, C., Recio-Totoro, B., Flores-Escobar, B., Lanz-Mendoza, H., Soberón, M., Bravo, A. 2015. Nitric oxide participates in the toxicity of Bacillus thuringiensis Cry1Ab toxin to Manduca sexta larvae. Peptides. 68: 134-139. 6. Gómez, I., Flores, B., Bravo, A., and Soberón, M. 2015. Bacillus thuringiensis Cry1AbMod toxin counters tolerance associated with low cadherin expression but not that associated with low alkaline phosphatase expression in Manduca sexta. Peptides. 68: 130-133 7. Zúñiga-Navarrete, F., Gómez, I., Peña, G., Amaro, I., Ortíz, E., Becerril, B., Ibarra, J. E., Bravo, A., Soberón, M. 2015. Identification of Bacillus thuringiensis Cry3Aa toxin Domain II loop 1 as the binding site of Tenebrio molitor cadherin repeat CR12. Insect Biochemistry and Molecular Biology. 59: 50-57 8. Monnerat, R., Martins, E., Macedo, C., Queiroz, P., Praça, L., Soares, C. M., Moreira, H., Grisi, I., Silva, J., Soberón, M., and Bravo, A. 2015 Evidence of field-evolved resistance of Spodoptera frugiperda to Bt corn expressing Cry1F in Brazil that is still sensitive to modified Bt toxins. Plos One. 10, e0119544. 9. Shu, C., Tan, S., Yin, J., Soberon, M., Bravo, A., Liu, C., Geng, L., Song, F., Li, K., Zhang, J. 2015. Assembling of Holotrichia parallela (Dark Black Chafer) midgut tissue transcriptome and identification of midgut proteins that bind to Cry8Ea toxin from Bacillus thuringiensis. Applied Microbiology and Biotechnology. 99: 7209-7218. 10. Tabashnik, B. E., Zhang, M., Fabrick, J. A., Wu, Y., Gao, M., Huang, F., Wei, J., Zhang, J., Yelich, A., Unnithan, G. C., Bravo, A., Soberón, M., Carrière, Y., Li, X. 2015. Dual mode of action of Bt proteins: protoxin efficacy against resistant insects. Scientific Reports. 5, 15107. 11. Pacheco, S., Cantón, E., Zuñiga-Navarrete, F., Pecorari, F., Bravo A., Soberón M. 2015 Improvement and efficient display of Bacillus thuringiensis toxins on M13 phages and ribosomes. AMB express. 5, 73. 12. Ocelotl, J., Sánchez, J., Arroyo, R., García-Gómez, B. I., Gómez, I., Unnithan, G. C., Tabashnik, B. E., Bravo, A., Soberón, M. 2015. Binding and Oligomerization of Modified and Native Bt Toxins in Resistant and Susceptible Pink Bollworm. Plos One. Aceptado-En prensa 13. Cantón, P. E., Cancino-Rodezno, A., Gill, S. S., Soberón, M., Bravo, A. 2015 Transcriptional cellular responses in midgut tissue of Aedes aegypti larvae following intoxication with Cry11Aa toxin from Bacillus thuringiensis. BMC Genomics. Aceptado-En prensa Capítulos en libros por invitación: 1. Soberón,M. Gomez,I. Garcia-Gomez,B.I. Carmona,D. Ocelotl,J. Villanueva,F. Flores,B. Bravo,A. 2014. Mode of action of mosquitocidal toxins from Bacillus thuringiensis and their use in control of insect vectors of human diseases en: Biotechnology: beyond borders. CSIR National Chemical Laboratory. pags. 279-288 2. Bravo,A. Martinez-de-Castro,D.L. Sanchez,J. Munoz-Garay,C. Matus,V. Canton,P.E. LopezDiaz,J. Portugal,L. Mendoza,G. Soberón,M. 2014. Mode of action of Bacillus thuringiensis toxins and their use in transgenic crops to control insect pests en: Biotechnology: beyond borders. CSIR National Chemical Laboratory. pags. 122-134 3. Bravo,A. Soberon,M. Gao,Y. 2015. Preface en: Bt Resistance: Characterization and Strategies for GM Crops Producing Bacillus thuringiensis Toxins. Oxford. CABI. ISBN-13: 9781780644370 pags. xi-xii 4. Bravo,A. Gomez,I. Mendoza,G. Gaytan,M. Soberon,M. 2015. Different models of the mode of action of 3d-Cry toxins from Bacillus thuringiensis (editores Bravo,A. Soberon,M. Gao,Y) en: Bt resistance – characterization and strategies for GM crops expressing Bacillus thuringiensis toxins. Oxford. CABI. ISBN-13: 9781780644370. Chapter 6, pp 56-69 5. Soberon,M. Garcia-Gomez,B.I. Pacheco,S. Sanchez-Quintana J Tabashnik,B.E. Bravo,A. 2015. Countering pest resistance with genetically modified Bt toxins. (editores Bravo,A. Soberon,M. Gao,Y) en: Bt resistance-characterization and strategies for GM crops expressing Bacillus thuringiensis. Oxford. CABI. ISBN-13: 9781780644370. Chapter 14, pp 150-162 6. Bravo,A. Martinez-de-Castro,D.L. Sanchez-Quintana J Canton,P.E. Mendoza,G. Gomez,I. Pacheco,S. Garcia-Gomez,B.I. Onofre,J. Ocelotl,J. Soberon,M. 2015. Mechanism of action of Bacillus thuringiensis insecticidal toxins and their use in the control of insect pests en: Alouf,J.E. Comprehensive Sourcebook of Bacterial Protein Toxins 4a ed. Elsevier. ISBN 9780128001882 Chapter 30, pags. 858-873 7. Grande-Cano, R. and Gómez, I. (2015) Identification and characterization of receptors for insecticidal toxins from Bacillus thuringiensis. In: Biocontrol of Lepidopteran pests. Soil Biology. Editores: K. Sowjanya Sree and Ajit Varma. Springer. Pags. 71-88. ISBN: 978-3-31914498-6 (Print) 978-3-319-14499-3 (Online) Libros editados: 1. Soberon,M. Gao,Y.L. Bravo,A. 2015. Bt resistance- characterization and strategies for GM crops expressing Bacillus thuringiensis toxins. Oxford:. CABI. Alumnos Graduados: Fernando Zúñiga Navarrete, Estudiante Doctorado (tutor MS, Octubre 2014) Alan I. Jiménez Reyes, Estudiante Maestría (tutor MS, Octubre 2015) Francisca Villanueva, Estudiante Maestría (tutor MS, Biviana Flores, Estudiante de Doctorado (tutor IG, Febrero 2014) Abril 2014) Donativos: 1. NIH National Institutes of Health. “Mechanism of mosquitocidal action of Bacillus thuringiensis” 2R01 AI066014-06 (Resp. Sarjeet S. Gill) sub-contrato 500,000.00 USD (grupos Alejandra Bravo y Mario Soberón). Julio 2010-Diciembre 2015 2. Convenio Pioneer Dupont 750,000.00 USD (grupos Mario Soberón y Alejandra Bravo) Enero 2010-Diciembre 2014. 3. CONACyT, Contrato No. 179977. El papel del transportador ABCC2 en el modo de acción de las toxinas Cry1A de Bacillus thuringiensis. $ 1,400,000.00 Enero 2013 tres años 4. PAPIIT-UNAM. No IN213514 (Responsible IG) $ 766,000.00 pesos. 2014-2016 5. DGAPA, UNAM Contrato IN201515. Estudio del mecanismo de resistencia de la palomilla de torso diamante a las toxinas Cry1A de Bacillus thuringiensis y estrategias moleculares para contender con la resistencia a estas toxinas. (Responsible MS) $780,000.00. 3 años. Enero 2014. 6. Convenio Pioneer Dupont 750,000.00 USD (grupos Mario Soberón y Alejandra Bravo) Enero 2015-Diciembre 2019. Patentes otorgadas: 1. Supression of resistance in insects to Bacillus thuringiensis Cry toxins, using toxins that do not require the cadherin receptor. Patente México No. 329408 2. Composición de un bioinsecticida para el control biólogico de larvas de mosquitos, vectores de enfermedades, con efectividad estabilizada. Instituto Mexicano de la Propiedad Intelectual. MX/a/20011/202100 Títulos y autores de los cárteles que presentará el grupo: 1. Study of Bacillus thuringiensis Cry1Ab and Cry1Ac protoxins interaction with cadherin-like receptor from Manduca sexta. Arlen Peña-Cardeña, Alejandra Bravo, Mario Soberón, Isabel Gómez 2. Eficiente despliegue de toxinas producidas por Bacillus thuringiensis en bacteriofagos filamentosos M13 y ribosomas. Sabino Pacheco, Emiliano Cantón, Fernando Zuñiga, Alejandra Bravo y Mario Soberón Grupo de Mario Zurita (2014-2015) Título: Drosophila, transcripción, cromatina y cáncer. Resumen: Drosophila es aún el mejor modelo de Biología del Desarrollo en animales. En el grupo trabajamos diferentes aspectos relacionados a la regulación de la expresión genética, la dinámica de la cromatina, la estabilidad del genoma durante el desarrollo y su relación con el cáncer. Nuestro principal modelo de trabajo es Drosophila melanogaster y recientemente hemos iniciado estudios en células humanas cancerosas en las que estamos aplicando algunas de la preguntas que tenemos la mosca. En cuanto a la regulación de la transcripción durante el desarrollo, por más de 15 años hemos trabajado con el factor de transcripción y reparación del DNA TFIIH. Con respecto a la dinámica de la cromatina estamos estudiando como esta responde al daño al DNA, así como el papel del remodelador de la cromatina ATRX en el desarrollo. Relacionado a esto también trabajamos con genes del grupo trithorax, en particular con tonalli que es una ligasa de SUMO. En cáncer estamos analizando el papel de TFIIH cuando una célula se transforma a cancerosa y como cambia la heterocromatina durante este proceso. En esta ocasión se presentarán ejemplos de algunos de estas líneas de investigación, en particular sobre la dinámica de TFIIH en el desarrollo temprano, en como la ausencia simultanea de TFIIH y p53 sobre activa a la kinasa de jun (JNK) para causar muerte celular y como cambia la heterocromatina cuando hay daño al DNA por radiación UV. Integrantes del Grupo: Mario Zurita (jefe de grupo) Martha Vázquez (inv. Ascociada) Viviana Valadez (inv. Asociada) Denhi Schnabel (inv. Asociada) Juan Manuel Murillo (Postdoc) Grisel Cruz (estudiante de doctorado) Alyeri Bucio (estudiante de doctorado) Cinthya Gurrión (estudiante de doctorado) Silvia Meyer (estudiante de doctorado) Maritere Urióstegui (estudiante de doctorado) Marco Antonio Rosales (estudiante de maestría) Sarai Valerio (estudiante de maestría) Yoselin Chávez (estudiante de maestría) Samanta Cruz (estudiante de LCG) Publicaciones: 1. Claudia Villicaña, Grisel Cruz, Mario Zurita. 2014. The basal transcription machinery as target for cancer therapy. Cancer Cell international 14:18 2. Brenda López-Falcón1*, Silvia Meyer-Nava1*, Benjamín Hernández-Rodríguez1, Adam Campos2, Daniel Montero1, Enrique Rudiño2, Martha Vázquez1, Mario Zurita1§, Viviana Valadez-Grahaml. 2014. Characterization of the Drosophila group ortholog to the N-terminal portion of the vertebrate Alpha-thalassemia and mental retardation X-linked (ATRX) protein. PLOS ONE (ec 1;9(12):e113182. doi: 10.1371/journal.pone.0113182). 3. Grisel Cruz, Mandy Juárez, Sarai Valerio-Cabrera and Mario Zurita*. 2015. Mitotic bookmarking by TFIIH during zygotic genome activation in Drosophila suggests the existence of a short-term transcriptional memory mechanism (sometido). 4. Grisel Cruz, Mandy Juárez, Viviana Valadez-Graham and Mario Zurita*. 2015.. TFIIH is required for male meiotic progression and cell differentiation in Drosophila spermatogenesis (sometido). Alumnos graduados: Maritere Urióstegui (Maestría). Donativos: 1. Rho y la localización del plasma germinal durante la ovogénesis de pez cebra PAPIIT Número: IN212514 (Denhi Schnabel). 2. Donativo 177393; CONACyT Joven Investigador (2013-2015) 3. Estudio de la proteína dAtrx en el mantenimiento telomérico de Drosophila melanogaster. (Viviana Valadez) 4. PAPIIT IN204915 (3 AÑOS) Viviana Valadez. Estudio de las proteínas ADD y su papel en la organización de la cromatina. 5. Conacyt #99654 "Identificación y caracterización de factores que interaccionen con TnaA, una proteína del grupo Trithorax en Drosophila melanogaster (2012-2014) Martha Vázquez. 6. Dgapa IN202613 " Perfiles de asociación a la cromatina de proteínas del grupo Trithorax y la sumoilación de una proteína del grupo polycomb en fondos defectuosos de la E3 ligasa de sumo, TnaA en Drosophila melanogaster" (2013-2015) Martha Vázquez. 7. DGAPA IN200315 " TFIIH en el inicio de la expresión genética en el embrión temprano y en la diferenciación celular en Drosophila melanogaster" (2015-2017) Mario Zurita 8. CONACYT #219673 "El papel del factor de transcripción y reparación del DNA TFIIH en mitosis, en la activación de la transcripción cigótica y la espermatogénesis en Drosophila melanogaster" (2015-2017) Mario Zurita 9. CONACYT #250588 - Infraestructura, " Implementación y equipamiento de un laboratorio de epigenética en el Instituto de Biotecnología de la UNAM" (2015) Mario Zurita Participación en docencia: Curso básico de biología molecular de la maestría en ciencias bioquímicas (2014). Divulgación: http://www.launion.com.mx/morelos/sociedad/noticias/80000-destaca-academia-mexicana-deciencias-investigaci%C3%B3n-que-desarrolla-acad%C3%A9mico-del-ibt-de-la-unam,-campusmorelos.html (M. Zurita) Academia Mexicana de Ciencias Boletín AMC/249/15 México, D.F., 21 de octubre de 2015 Estudian defectos en la reparación del ADN en distintas enfermedades. Títulos y autores de los carteles que presentará el grupo: 1. Determinación del mecanismo de traducción de un mRNA bicistrónico en Drosophila melanogaster. Alyeri Bucio 2. Knocking down TnaA, a trxG putative SUMO E3 ligase. Marco Antonio Rosales Participantes Jueves 10 de Diciembre del 2015 Grupo de Gustavo Pedraza Alva (2014-2015) Consorcio de Neuroinmunobiología Título: El Yin Yang del sistema inmune y el sistema nervioso. Resumen: La visión clásica de que un proceso inflamatorio se iniciaba solo por la activación de células del sistema inmune en respuesta a patógenos o al daño tisular, ha cambiado en los últimos años. Ahora sabemos que este proceso inflamatorio lo puede activar prácticamente cualquier célula del organismo y que también se inicia en respuesta a alteraciones metabólicas. Recientemente, se ha definido que muchos de las señales de daño derivadas de alteraciones en el metabolismo (colesterol, lípidos, acido úrico, niveles elevados de glucosa, etc.) inducen, por diferentes mecanismos, el ensamblaje del inflamasoma, con la consecuente activación de caspasa-1 y la producción de IL-18, iniciando así un proceso inflamatorio, que ante la continua presencia del estimulo se perpetua, alterando la homeostasis del organismo. En este contexto, discutiré los avances del proyecto encaminado a elucidar los mecanismos moleculares mediante los cuales la inflamación crónica en el sistema nervioso central (SNC) promueve el deterioro cognitivo en animales modelo de la enfermedad de Alzheimer. Además de entender como la inflamación crónica afecta las funciones del SNC, en el consorcio de neuroinmunobiología también estamos interesados en entender como el SNC modula procesos inflamatorios crónicos y agudos en la periferia. En esta ocasión, presentare evidencias experimentales que indican que una estimulación somatosensorial (Ambiente Enriquecido) modula negativamente la colitis experimental. Integrantes del Grupo: Administrativos Clara Maritza Díaz Aldama María del Carmen Gante Villa Rubén Blancas Naranjo Académicos Dr. Tomás Villaseñor Toledo. Investigador Asociado Oswaldo López Gutiérrez. Técnico Académico Posdoctorados Dr. Tomás Villaseñor Toledo Estudiantes Nilda del Carmen Sánchez Castellanos Jonathan Salazar León Ana Laura Valdez Hernández Edgardo Madrid Paulino Rafael Alejandro Maldonado Bravo Erick Israel Pérez Garcia Alejandro Ramírez Olvera Alejandro Costet Mejía Jorge Luis Almazán Ochoa Publicaciones: 1. Pérez-García E.I., Meza-Sosa K.F., López-Sevilla Y, Camacho-Concha N., Sánchez N.C., Pérez-Martínez L., Pedraza-Alva, G. 2015. Merlin negative regulation by miR-146a promotes cell transformation. BBRC, DOI 10.1016/j.bbrc.2015.10.156. 2. Pedraza-Alva G, Pérez-Martínez L, Valdez-Hernandez L, Meza-Sosa KF, Ando-Kuri M. 2015 Negative regulation of the inflammasome: keeping inflammation under control. Immunological Reviews. 265: 231-257. 3. Meza-Sosa KF, Pérez-García EI, Camacho-Concha N, López-Gutiérrez O, Pedraza-Alva G, Pérez-Martínez L. (2014) MiR-7 promotes epithelial cell transformation by targeting the tumor suppressor KLF4. PLoS One. 9(9):e103987. doi:10.1371/journal.pone.0103987. 4. Ferat-Osorio, E., Sánchez-Anaya, A., Gutiérrez-Mendoza, M., Boscó-Gárate, I., Wong-Baeza, I., Pastelin-Palacios, R., Pedraza-Alva, G., Bonifaz, L.C., Cortés-Reynosa, P., Pérez-Salazar, E., Arriaga-Pizano, L., López-Macías, C., Rosenstein, Y., Isibasi, A. 2014. Heat shock protein 70 down-regulates the production of toll-like receptor-induced pro-inflammatory cytokines by a heat shock factor-1/constitutive heat shock element-binding factor-dependent mechanism. J Inflamm. 11:19. doi: 10.1186/1476-9255-11-19. 5. Meza-Sosa, K.F., Pedraza-Alva, G. and Pérez-Martínez, L. 2014. microRNAs: key triggers of neuronal cell fate. Front Cell Neurosci. 8:175. doi: 10.3389/fncel.2014.00175. Alumnos graduados: 1. Rafael Alejandro Maldonado Bravo. Facultad de Ciencias Biológicas. Universidad Autónoma del Estado de Morelos. Evaluación de la participación de la vía de PKC en la inducción de los flujos de calcio producidos por Mycobacterium bovis en macrófagos. Junio 2014. 2. Erick Israel Pérez Garcia. Facultad de Ciencias. Universidad Nacional Autónoma de México. Efecto de la Inhibición del supresor de tumores Merlin por medio de la sobreexpresión de los microRNAs 7 y 146a, durant el proceso de transformación celular. Octubre 2014. Donativos: 2012-2014CONACYT. 154542: El papel del inflamasoma en la neurodegeneración inducida por la acumulación de péptidos -Amyloides. 2012-2015 ICGEB. CRP/MEX11-01: Characterization of the transduction pathways activated by M. Tuberculosis leading to inefficient immune response. 2013-2015 DGAPA/UNAM. IN209513: El papel del inflamasoma de Nalp1b1 en la resistencia a insulina y leptina resultantes del proceso inflamatorio asociado a la obesidad. Divulgación: Pedraza-Alva, G. Torres-Huerta, A. Rosenstein, Y. 2015. Las intrincadas redes de la inmunidad Ciencia, Revista de la Academia Mexicana de Ciencia, 66, 27-35. Participación en docencia: 1. Curso de Biología Celular. Licenciatura en Ciencias Genómica, UNAM. 2. Taller “La Biología a través de las biomoléculas: nuevos paradigmas y aplicaciones”. Facultad de Ciencias, UNAM. 3. Curso de Biología Celular. Maestría y Doctorado en Ciencias Bioquímicas, UNAM. Títulos y autores de los carteles que presentará el grupo: 1. Caracterización de las vías de transducción de señales activadas por M. bovis que favorecen el proceso de infección y su sobrevivencia en el macrófago. R. Maldonado, E. Madrid y T. Villaseñor, L. Pérez y G. Pedraza. 2. MicroRNAs: En transformación celular, diferenciación y disfunción neuronal. E. García y N. Sánchez, J. López, A. Urban, S. Díaz de León, L. Pérez y G. Pedraza 3. Un mismo inflamasoma y dos estímulos, respuestas opuestas? J. Salazar, L. Valdez, M. Pedraza, L. Pérez y G. Pedraza. Grupo de Octavio Tonatiuh Ramírez Reivich (2014-2015) Título: Escalamiento descendente: Estudio del efecto de gradientes de pH en cultivos de E. coli recombinate sobre la producción de ADN plsmídico. Resumen: El escalamiento descendente es un acercamiento conceptual que ofrece herramientas experimentales para estudiar, a nivel laboratorio y de manera práctica, eficiente y económica, condiciones medioambientales que típicamente se presentan en biorreactores de gran escala. En particular, el escalamiento descendente es de gran utilidad para determinar el efecto de heterogeneidades ambientales, tales como oxígeno disuelto y concentración de substratos, sobre el metabolismo y fisiología celular, así como sobre el desempeño de un bioproceso particular. Los gradientes ambientales que se pueden presentar en biorreactores de gran escala generalmente son el resultado de limitaciones físicas o restricciones inherentes en el diseño de sistemas de producción a nivel industrial. Una variable particularmente importante es el pH, ya que en biorreactores de gran escala se pueden presentar gradientes, de hasta varias unidades, entre las zonas de adición de álcali y/o ácido y zonas bien mezcladas del seno del líquido. A la fecha, existen pocos estudios de escalamiento descendente del pH debido a la dificultad de oscilar continuamente esta variable sin generar un problema de formación de sales y aumento de osmolaridad. E. coli es uno de los hospederos mas empleados alrededor del mundo para producir proteínas recombinantes, siendo imprescindible no sólo en aplicaciones biotecnológicas sino también en estudios básicos de muchas disciplinas. Consecuentemente, la información existente sobre fisiología y metabolismo de este microorganismo es realmente extensa. No obstante, un tema de gran importancia pero muy poco estudiado es el comportamiento de E. coli recombinante productora de ADN plasmídico al ser expuesta a fluctuaciones medioambientales, particularmente de pH. En el presente trabajo se diseñó un sistema de escalamiento descendente, constituido por dos biorreacotres de tanque agitado interconectados, cada uno con control de pH, independiente y diferente, y entre los que se recirculó caldo de cultivo para simular tiempos de residencia entre 60 y 240 s típicos de biorreactores de gran escala. En cultivos por lote fue posible generar gradientes de pH hasta de 0.9 unidades, con respecto a la condición control de 7.0, y determinar su efecto en parámetros cinéticos y estequiométricos, así como cambios en la topología del ADN plasmídico producido. Asimismo, se determinó el efecto de la respuesta transcripcional de 10 genes relacionados al estrés alcalino, incluyendo varios relacionados con el transporte de iones, el catabolismo de aminoácidos y reguladores transcripcionales. Se mostrará cómo E. coli recombinante responde de manera diferencial entre el estrés alcalino constante y el intermitente. Los resultados obtenidos son de utilidad para el diseño de nuevas cepas bacterianas que puedan contender mejor con el problema de gradientes de pH, así como para desarrollar mejores diseños de biorreacotres y estrategias de operación de cultivos a gran escala. Integrantes del Grupo: O.Tonatiuh Ramírez, Laura A. Palomares, Vanessa Hernández, Ana Ruth Pastor, Martha Alicia Contreras, Rosa Román, Larisa Campos, Karin Levy, Xóchitl González, Juana Ferrer, Antonio Dorantes, Esteban Peguero, Mabel Rodríguez, Oriana Niño, José Tonatiuh Cortes, Alberto Porras, Alejandra Mejía, Andrea Téllez, Carolina Gómez, Cinthia Martínez, David Hidalgo, David S. Moreno, Dayanira Paniagua, Eduardo Ramírez, Elizabeth Carrasco, Enrique Paz, Fernando Aponte, Francisca Villanueva, Gonzalo González, Hernán Ortiz, Iris Bravo, Israel Montes de Oca, Jaqueline Zuñiga, Jocelyn Uribe, Jonathan Arredondo, Karina E. García, Karina Espíndola, Laura Tapia, Lucía Cabrera, Mabel Rodríguez, Norman Brito, Ramón Carrasco, Ricardo Castro, Ulises Barrón, Uri Mendoza, Victor López, Violeta Guadarrama, Wendy García. Publicaciones: 1. M. Wunderlich, H. Taymaz-Nikerel, G. Gosset, O. T. Ramírez, A. R. Lara. 2014. Effect of Growth Rate on Plasmid DNA Production and Metabolic Performance of Engineered Escherichia coli strains" Journal of Bioscience and Bioengineering, 117(3), 336-342. 2. W.A. Rodríguez-Limas, A.R. Pastor, E. Esquivel-Soto, F. Esquivel-Guadarrama, O. T. Ramírez, LA Palomares. 2014. Immunogenicity and Protective Efficacy of Yeast Extracts Containing Rotavirus-Like Particles: A Potential Veterinary Vaccine”, Vaccine, 32, 2794-2798. 3. A. R. Pastor-Flores, WA Rodríguez-Limas, M. A. Contreras, E Esquivel-Soto, F. EsquivelGuadarrama, O. T. Ramírez, L.A.Palomares. 2014. The Assembly Conformation of Rotavirus VP6 Determines its Protective Efficacy Against Rotavirus Challenge in Mice”, Vaccine, 32, 2874-287. 4. L. Carreño-Fuentes, G. Plascencia-Villa, L. A. Palomares, S. E. Moya, O. T. Ramírez. 2014. Modulating the physicochemical and structural properties of gold-functionalized protein nanotubes through thiol surface modification, Langmuir, 30: 49, 14991 – 14998. 5. R.M. Castro Acosta, W.A. Rodríguez Limas, Brenda Valderrama, O.T. Ramírez, L.A. Palomares. 2014. Effect of Metal Catalyzed Oxidation in Recombinant Viral Protein Assemblies, Microbial Cell Factories, 13:25. 6. G.Plascencia-Villa, L. Carreño-Fuentes, D. Bahena, M.J. Yacamán, L.A. Palomares and O.T. Ramírez. 2014. Characterization of Conductive Nanobiomateriales Derived from Viral Assemblies by Low-Voltage STEM Imaging and Raman Scattering Nanotechnology, 25, 385706 (11pp). 7. M. Rodríguez, Ch. Wood, R. Sánchez, R.M. Castro Acosta, O.T. Ramírez, L.A. Palomares. 2014. Understanding Internalization of Rotavirus VP6 Nanotubes by Cells: Towards a Recombinant Vaccine” Archives in Virology, 159, 1005-1015. 8. J. T. Cortés, N. Flores, F. Bolívar, A. R. Lara, and O. T. Ramírez. 2015. Physiological effects of pH gradients on Escherichia coli during plasmid DNA production”, Biotechnology and Bioengineering, Aug 24 [Epub ahead of print], 8 SEP 2015, DOI: 10.1002/bit.25817. Alumnos Graduados: Doctorado 1. M. en C. Liliana Carreño;; “Funcionalización Dirigida y Localizada de Nanotubos de VP6 de Rotavirus”. Fecha de titulación: 18 marzo 2014 Maestría 2. José Tonatiuh Cortés Esquivel “Estudio de la respuesta metabólica de E. coli bajo estrés inducido por pH en una cepa productora de ADN plasmídico” Programa en Ciencias Bioquímicas. Fecha de titulación: 8 diciembre 2014. 3. Elizabeth Carrasco Caballero “Efecto de las estrategias de termo-inducción sobre porducción de proteína recombinante y la composición de los cuerpos de inclusion de E. coli” Programa en Ciencias Bioquímicas. Fecha de titulación: 20 febrero 2015 En proceso 4. David Hidalgo Vázquez;; “Diseño de un Microsistema de Cultivo para Exponer Células de Eucariotes Superiores a Condiciones de Estrés Típicas de un Bioproceso”, Programa en Ciencias Bioquímicas de enero 2013 a la fecha. 5. Hernán Alejandro Ortiz Maldonado; “Diseño y desarrollo de un nuevo sistema de termoinducción” Programa en Ciencias Bioquímicas de agosto 2013 a la fecha. 6. Wendy Ivone García García: Estudio de la interacción de nanotubos de VP6 de rotavirus funcionalizados con metales y células en cultivo: Hacia nuevas aplicaciones” Programa en Ciencias Bioquímicas de enero 2014 a la fecha. 7. Otilia Lucía Cabrera Castro; “Efecto del tiempo de residencia de un anticuerpo monoclonal en el sobrenadante de cultivo de células CHO, sobre su patrón de glicosilación”, Programa en Ciencias Bioquímicas de enero 2015 a la fecha. Donativos: 1. Diseño de Biorreactores para Escalamiento Descendente para el Estudio de Células Individuales. Responsable: Dr. O.T. Ramírez / Financiado por DGAPA IT-201214-2 (enero 2014 – diciembre 2015). 2. Construcción y equipamiento de un laboratorio de análisis de medicamentos biotecnológicos en el Estado de Morelos. Responsable: Dr. O.T. Ramírez / Fondo Mixto de Fomento a la Inversión Científica y Tecnológica CONACYT-Gobierno Libre y Soberano de Morelos. FOMIX-249789 3. Donativos varios con diversas empresas. Transferencias Tecnológicas: 1. Desarrollo y optimización de un clon y determinación preliminar de su biocomparabilidad con un producto de referencia. Transferida a la empresa “Laboratorios Liomont S.A. de C.V.” Convenio firmado el 31 de marzo 2014. Convenio DGAJ-SPI-220713-681 entre el Instituto de Biotecnología de la UNAM y Laboratorios Liomont S.A. de C.V. 2. Desarrollo y optimización de un anticuerpo monoclonal. Transferida a la empresa “Laboratorios Liomont S.A. de C.V.” Convenio firmado el 31 de marzo 2014. Convenio DGAJDPI-070514-856 entre el Instituto de Biotecnología de la UNAM y Laboratorios Liomont S.A. de C.V. Participación en docencia: 1st Course on Technological Platforms for Glycosciences: Glycosciences and Health Network; “Importance of glycoproteins and glycoprotein characterization for pharmaceuticals”, UAEM Cuernavaca Mor. 26 octubre 2015 Divulgación: 1. Medicamentos Biocomparables. Mesa redonda, Instituto de Biotecnología/ UNAM. Enero 2015 2. Valor agregado a productos de marca: Desarrolla Biotecnología fármacos biocomparables. Gaceta UNAM febrero 2015. 3. Logro del Instituto de Biotecnología: Vacuna recombinante contra rotavirus bovino, Gaceta UNAM 23 febrero 2015. 4. Ciencia Básica; Biotecnología, entidad con más patentes, Gaceta UNAM 17 marzo 2015. 5. Academia de Ciencias de Morelos/ Comunicado de Prensa: Habilitan a Unidades Científicas para asegurar medicamentos biotecnológicos en México. 1 abril 2015 6. Acuerdo con la Cofepris: La UNAM realizará pruebas a medicamentos, Gaceta UNAM 13 abril 2015 Ya cuenta con una Patente: Desarrolla Biotecnología cepas microbianas mas resistentes, Gaceta UNAM 23 abril 2015. 7. Entrevista para la revista Negocios “Mexican Biotechnology: Applied Technology for the improvement of life” agosto 2015. http://www.promexico.gob.mx/documentos/revistanegocios/pdf/ago-2015.pdf Títulos y autores de los carteles que presentará el grupo: 1. Efecto de las estrategias de termoinducción sobre la producción de proteína recombinante en Escherichia coli. Elizabeth Carrasco, O. Tonatiuh Ramírez. 2. Desarrollo de estrategias de alimentación para incrementar la producción de partículas pseudovirales de rotavirus en Saccharomyces cerevisiae. Martha A. Contreras, Ramón Carrasco, Ruth Pastor, Octavio T. Ramírez y Laura A. Palomares. Grupo Patricia León (2014-2015) Titulo: La regulación de cloroplastos y su influencia en el desarrollo de la planta Resumen: Una de las características más notables de las plantas es la presencia de organelos smiautónomos llamados plástidos, que desempeñan funciones vitales, incluyendo la fotosíntesis, la síntesis de metabolitos secundarios y de hormonas. Desde su adquisición, los plástidos han coevolucionado con la célula vegetal generando una intricada comunicación. A pesar de que los plastidos contienen un propio genoma, este codifica para menos un 5% de la proteínas requeridas para su función. La mayoría de las proteínas plastídicas están codificadas en el núcleo, incluyendo aquellas involucradas en la expresión del genoma plastídico. De esta manera el núcleo regula la función y desarrollo de los plástidos. Por su parte los plastidos también generan señales que modulan la expresión de genes nucleares y con las cuales comunican su estado metabólico y de desarrollo a la planta, a lo que se conoce como señalización retrógrada. Esta señalización es fundamental para coordinar de manera adecuada los cambios en los requerimientos y el desarrollo de los plástidos, con la expresión de las proteínas plastidicas codificadas en el núcleo. Un interés fundamental de nuestro grupo es entender los mecanismos de la intercomunicación entre plastidos y el núcleo. Con esta idea hemos identificado mutantes incapaces de poder tener un desarrollo adecuado de los organelos y de la planta. El análisis de estas mutantes ha permitido conocer aspectos novedosos de la regulación de plastidos y de su impacto en el desarrollo de la planta. En el caso de la regulación nuclear, hemos demostrado la participación de miembros de una familia muy numerosa de proteínas (codificadas en el núcleo) de interacción de RNA llamadas PPR. Estas proteínas son importadas a plastidos, donde tienen funciones centrales en la regulación postranscripcional plastídica. Algunas de ellas son elementos esenciales de la edición de transcritos esenciales de los plástidos. La edición de RNA es un mecanismo de regulación que ocurre frecuentemente en plastidos y mitocondrias y donde se modifica la secuencia de un transcrito en un sitio específico de C a U. Este proceso es vital para la correcta expresión de alrededor de 35 proteínas plastídicas. De manera interesante las proteínas PPRs son centrales para el reconocimiento específico del sitio de edición. Nuestros estudios también han aportado información de cómo las proteínas PPRs reconocen su sitio blanco de manera específica y de la interacción con otras proteínas del editosoma. Por otro lado, en los últimos años nuestro trabajo también se ha enfocado en la caracterización de señales generadas por los plastidos y como dichas señales influyen en la expresión nuclear y el desarrollo de la planta. A través de la caracterización de la mutante clb5, hemos generado evidencias de la existencia de una señal que afecta la expresión de genes cloroplásticos y nucleares y el desarrollo de la hoja. Esta señal se genera a partir de la acumulación de carotenos lineales como fitoflueno o zeta carotenos, como resultado de una disminución de los niveles de la enzima zeta caroteno desaturasa (ZDS). Aunque la estructura exacta de esta señal es aún desconocida, sabemos que es un nuevo apocaroteno generado a través de la enzima digoxigenasa de carotenos 4 (CCD4). Estudios de expresión de ZDS han mostrado que la expresión de DXS sigue un gradiente durante del desarrollo de la hoja. Finalmente nuestros datos recientes a través de análisis globales de expresión han mostrado el impacto que esta señal tiene sobre la expresión nuclear apoyando un papel importante en el desarrollo de plantas. Integrantes del Grupo: Adscritos Elizabeth Cordoba y Arturo Guevara Técnico Maricela Ramos Postdoctoral Cynthia Romero Doctorado Alma Hernández, Marel Chenge, Luis de Luna y Jesús López-Bucio Maestría: Medina, Julio Sierra, María Cruz, José Manuel González Licenciatura Joselín Palmira Domitzel Zagal Ricardo Eslava Ceclia Lara Carlos Villaseñor Dagmara Isabel García Yessica Gabriela Publicaciones: 1. López-Bucio J.S., Dubrovsky J.G., Raya-González J., Ugartechea-Chirino Y., López-Bucio J., de Luna-Valdez L.A., Ramos-Vega M., León P., Guevara-García A.A. 2014. Arabidopsis thaliana mitogen-activated protein kinase 6 is involved in seed formation and modulation of primary and lateral root development. 65: 169-183 J Exp Bot. doi:pii: S0278-6915(13)00748-5. ISSN 0022-0957. 2. Avendaño-Vázquez, A., Cordoba, C., Llamas, E., San Román, C., Nisar, N., De la Torre, S., Ramos-Vega, M., Gutiérrez-Nava, L., Cazzonelli, C.I., Pogson, BJ., León, P. 2014. An uncharacterized apocarotenoid-derived signal generated in zeta-carotene desaturase mutants regulates leaf development and the expression of chloroplast and nuclear genes in Arabidopsis. Plant Cell 26: 2524–2537. ISSN: 1040-4651. 3. de Luna-Valdez, L. A., Martínez-Batallar, A. G., Hernández-Ortiz. M., Encarnación-Guevara. S., Ramos-Vega, M., López-Bucio, J. S., León, P., Guevara-García, A. A. 2014. Proteomic analysis of chloroplast biogenesis (clb) mutants uncovers novel proteins potentially involved in the development of Arabidopsis thaliana chloroplasts. Journal of Proteomics 111: 148-164. ISSN: 1874-3919. 4. de Luna-Valdez, L.A., Martinez-Batallar, A.G., Hernandez-Ortiz, M., Encarnacion-Guevara, S., Ramos-Vega, M., Lopez-Bucio, J.S., Leon, P., Guevara-Garcia, A.A. 2014. Data for proteomic analysis of chloroplast biogenesis (clb) mutants uncovers novel proteins potentially involved in the development of Arabidopsis thaliana chloroplasts Data in Brief 1: 15-18. ISSN: 2352-3409 http://dx.doi.org/10.1016/j.did.2014.07.001 5. E. Cordoba, E., Aceves-Zamudio, D.L., Hernández-Bernal, A.F., Ramos-Vega M.G., León P. 2015. Sugar regulation of the SUGAR TRANSPORTER 1 (STP1) expression in Arabidopsis thaliana. Journal of Experimental Botany 66: 147-159. ISSN 0022-0957. 6. M. Ramos-Vega, A. Guevara-García, E. Llamas, N. Sánchez-León, V. Olmedo-Monfil, J. P. Vielle-Calzada and P. Leon (2015) Functional analysis of the CHLOROPLAST BIOGENESIS 19 PPR editing protein. New Phytology DOI: 10.1111/nph.13468. 7. de Luna Valdez, L. A., León Mejía P., Encarnación Guevara, S., Guevara-García, A. A. (2015) Chloroplast omics. In: Barh Delmya (Ed). Plant omics. Springer Verlag, BerlínHeidelberg, Alemania. Alumnos Graduados: Gabriela Medina, Joselin Palmira, María Cruz y Carlos Villaseñor. Donativos: DGAPA (2014-2016). Responsable Patricia León CONACYT (2015-2017). Responsable Patricia León DGAPA (2014-2016). Responsable Arturo Guevara CONACYT (2013-2015). Responsable Arturo Guevara DGAPA (2013-2015). Responsable Elizabeth Cordoba CONACYT (2013-2015). Responsable Elizabeth Cordoba Participación en docencia: Biología Celular, Biología Vegetal y Taller Facultad de Ciencias. Títulos de los carteles que presentará el grupo: 1. Una cascada de MAP cinasas está involucrada en el desarrollo del(a) embrión/semilla de Arabidopsis thaliana. 2. Identification of DNA binding proteins to the regulatory region implicated in the STP1 (SUGAR TRANSPORTER PROTEIN 1) gene sugar regulation in Arabidopsis thaliana. Grupo de Baltazar Becerril (2014-2015) Título: Avances en la generación de un anti-veneno recombinante de origen humano contra la picadura de alacranes ponzoñosos de México. Resumen: El carácter poli-clonal de los anti-venenos comerciales (obtenidos por inmunización de caballos con venenos de alacranes), contrasta con los logros de las investigaciones dedicadas a desarrollar nuevos antídotos. Mientras que el anti-veneno comercial va dirigido contra todos los componentes de los venenos (tóxicos y no tóxicos), las versiones modernas estarán compuestas de anticuerpos específicos dirigidos exclusivamente contra los componentes tóxicos del veneno. Para evitar el uso de animales en la producción de los anti-venenos y mejorar su eficiencia terapéutica, buscamos desarrollar un anti-veneno innovador compuesto de fragmentos de anticuerpos neutralizantes de origen humano en formato de cadena única (scFv; dominios variables de las cadenas pesada y ligera unidas por un péptido conector), los cuales al ser de menor tamaño que los correspondientes al anti-veneno de origen equino, muestran una mayor penetración y distribución en el organismo, lo cual en sí mismo representa una ventaja ante la rapidez con la que difunde el veneno. La estrategia experimental que hemos seguido para generar el nuevo anti-veneno, ha integrado el despliegue en fagos de bibliotecas de scFvs humanos, el tamizado racional de las mismas, la identificación y caracterización de variantes que reconocen a las principales toxina, además de la implementación de procesos de maduración de la afinidad de los scFvs. Resultados Previos: Del banco de scFvs humanos se aislaron dos scFvs parentales (3F y C1) que reconocen a la toxina más abundante y letal (Cn2) del alacrán más venenoso de México (Centruroides noxius). Ambos scFvs la reconocen, pero no la neutralizan, por lo que fueron madurados. De estos procesos, se obtuvieron los scFvs LR y RU1derivados de 3F y C1 respectivamente. Cada uno de ellos neutraliza a la toxina Cn2 y al veneno completo. Su administración simultánea en ratones, se tradujo en una protección mejorada ante un reto con 3 LD50 de veneno fresco, observándose una rápida recuperación. La estructura del complejo ternario (scFv LR-Cn2-scFv RU1), permitió identificar sus áreas de interacción con la toxina, correspondiendo a dos sitios independientes. Resultados destacados: La alta homología de las toxinas de los Centruroides, está relacionada con los diversos niveles de reactividad cruzada de los scFvs generados. LR y RU1, neutralizan a 5 y 3 toxinas diferentes de especies distintas, respectivamente. El conjunto de la estructura del complejo ternario y las diversas herramientas para implementar ingeniería de anticuerpos, permitió modular el reconocimiento y la afinidad de algunos scFvs hacia diferentes toxinas. Se identificaron residuos clave involucrados en la ampliación de la reactividad cruzada de scFvs derivados de RU1. La propiedad intelectual sobre estos resultados, fue solicitada recientemente. Esta línea que estamos siguiendo, nos permite anticipar la generación de un anticuerpo de tipo “universal” que pueda neutralizar un mayor número de toxinas. México, al ser un país bio-diverso, cuenta con un número significativo de especies tóxicas de alacrán. En los inicios del proyecto, había 7 especies tóxicas reportadas y actualmente son 14. Los avances significativos del proyecto en los últimos meses, han resultado en scFvs capaces de neutralizar a las toxinas principales y a los respectivos venenos de las especies: C. noxius, C. limpidus, C. suffusus, C. scupturatus, C. infamatus, C. hirsutipalpus y a las toxinas principales de C. tecomanus, C. elegans y C. ornatus, con el empleo de únicamente dos scFvs. Las perspectivas del proyecto se centran en la generación de nuevos scFvs capaces de neutralizar a las toxinas y venenos restantes. Se definirá cuales scFvs conformarán el nuevo antiveneno equivalente al comercial. Finalmente, habrá que escalar la producción, bajo buenas prácticas de manufactura, de los scFvs seleccionados para entonces implementar su formulación y hacer pruebas pre-clínicas. Integrantes del Grupo: Investigadora Asociada contratada por honorarios y actual Catedrática CONACyT, la Dra Lidia Riaño Umbarila Técnicos Académicos: Dr. Ernesto Ortiz, M. en C. Timoteo Olamendi, M. en C. Leopoldo Güereca, Biol. Rosalba Sánchez. Estudiantes: Licenciatura: Selene Jocelyn Uribe Romero. Maestría: Guillermo Fernández Taboada, Jonathan Noé Arredondo López y Mónica García-Montelongo. Doctorado: Miryam Ivette Villalba Velázquez, Oscar Daniel Luna Martínez y Everardo Remi Rodríguez Rodríguez Publicaciones: 1. Villalba, M.I., Canul-Tec, J.C., Luna-Martinez, O.D., Sanchez-Alcala, R., Olamendi-Portugal, T., Rudino-Pinera, E., Rojas, S., Sanchez-Lopez,R., Fernandez-Velasco,D.A., Becerril,B. 2015. Site-directed Mutagenesis Reveals Regions Implicated in the Stability and Fiber Formation of Human lambda3r Light Chains J. Biol Chem, 290, 2577-2592. 2. Quintero-Hernandez,V. Ramirez-Carreto,S. Romero-Gutierrez,M.T. Valdez-Velazquez, L.L. Becerril,B. Possani, L.D. Ortiz,E. 2015. Transcriptome Analysis of Scorpion Species Belonging to the Vaejovis Genus PLoS ONE, 10, e0117188. 3. Zuñiga-Navarrete, Fernando; Gómez, Isabel; Peña, Guadalupe; Amaro, Itzel; Ortiz Suri, Ernesto; Becerril, Baltazar; Ibarra, Jorge; Bravo de la Parra, Maria Alejandra; Soberón Chávez, Mario. (2015). Identification of Bacillus thuringiensis Cry3Aa toxin Domain II loop 1 as the binding site of Tenebrio molitor cadherin repeat CR12. 2015. Insect Biochemistry and Molecular Biology, 59, 50-57. 4. Ramirez-Carreto, S., Jimenez-Vargas,J.M., Rivas-Santiago, B., Corzo, G., Possani, L.D., Becerril,B., Ortiz, E. (2015). Peptides from the scorpion Vaejovis punctatus with broad antimicrobial activity Peptides, 73, 51-59. 5. Lidia Riano-Umbarila, Luis M. Ledezma-Candanoza, Hugo Serrano-Posada, Guillermo Fernandez-Taboada, Timoteo Olamendi-Portugal, Sonia Rojas-Trejo, Ilse V. Gomez-Ramirez, Enrique Rudino-Pinera, Lourival D. Possani, and Baltazar Becerril (2016). Optimal Neutralization of Centruroides noxius Venom Is Understood through the Structural Complex between Two Antibody Fragments and the Cn2 Toxin. J. Biol. Chem. (en prensa). 6. Timoteo Olamendi-Portugal, Adam Bartok, Fernando Zamudio-Zuñiga, Andras Balajthy, B. Baltazar Becerril, Gyorgy Panyi, and Lourival Domingos Possani. Isolation, chemical and functional characterization of several new K+-channel blocking peptides from the venom of the scorpion Centruroides tecomanus. Toxicon (sometido) Capítulos en libros: Rodriguez Rodriguez, Everardo Remi., Lidia Riaño Umbarila., Lourival D. Possani, Baltazar Becerril. 2014. Anti-venoms: Recombinant neutralizing antibodies, a new generation of antivenoms. Gopalakrishnakone, P et al (eds). Springer,, pp 139-157. Del Pozo, L., Becerril, B., Ochoa-Leyva, A., Rodríguez-Ambríz, S.L., Pérez-Carreón, J.I., Zavala-Padilla, G., Sanchez-López, R and Fernández-Velasco, D.A. The structural determinants of the immunoglobulin light amyloid aggregation. L. Olivares-Quiróz et al. (eds). Springer, 2015., pp 1-28 Alumnos Graduados: Para la obtención del título de Licenciado en Ciencias (Facultad de Ciencias). Universidad Autónoma del Estado de Morelos. Evaluación de la reactividad cruzada y capacidad neutralizante de fragmentos variables de cadena sencilla contra toxinas de venenos de alacranes mexicanos. Presentada por Selene Jocelyn Uribe Romero, 6 de febrero del 2015. La Dra. Lidia Riaño Umbarila fue la tutora principal. Maestría en Ciencias Bioquímicas: Caracterización de variantes del scFv C1 contra toxinas de alacranes Mexicanos. Presentada por Guillermo Fernández Taboada el 27 de junio de 2014. La Dra. Lidia Riaño Umbarila fue la asesora técnica de esta tesis. Maduración de la afinidad del scFv RU2 hacia la toxina Cll2 del veneno del alacrán Centruroides limpidus limpidus Karsch. Presentada por Jonathan Noé Arredondo López el 13 de mayo de 2015. La Dra. Lidia Riaño Umbarila fue la asesora técnica de esta tesis. Despliegue en fagos de variantes del amino terminal de CssII. Presentada por Mónica GarcíaMontelongo el 25 de junio del 2015. El Dr. Ernesto Ortíz fue el Tutor. Maestría en Medicina Molecular. Facultad de Medicina UAEM: Generación de scFvs capaces de neutralizar a la toxina Cll2 del alacrán Centruroides limpidus limpidus. Presentada por la Biol. Ilse Viridiana Gómez Ramírez el 20 de noviembre de 2015. La Dra. Lidia Riaño fungió como asesora técnica de la tesis. Doctorado en Ciencias Bioquímicas: Regiones implicadas en la estabilidad termodinámica y formación de fibra de la cadena ligera λ3r humana y su relación con la amiloidosis AL. Presentada por Miryam Ivette Villalba Velázquez el 7 de agosto de 2015. Donativos: CONACYT CB 2010. Proyecto 155099, 1´556, 755 pesos para 3 años, finalizó en febrero del 2015. PAPIIT IN 201913, $760.OOO durante el período 2013-2015. CONACYT 266067 (CATEDRAS), 500,000 para el año 2015. CONACYT 246924 (PROBLEMAS NACIONALES), 3´600,000 durante el periodo 2015-2016 Participación en docencia: Cursos de Biología Molecular, Métodos Analíticos en Biotecnología y Bases fundamentales para el estudio de las interacciones lípido-proteína del Posgrado en Ciencias Bioquímicas. Curso de Avances Genómicos de la Licenciatura en Ciencias Genómicas. Divulgación: Becerril, B, L. Riano, L.D. Possani. 2014. Bancos de anticuerpos recombinantes de origen humano: una fuente ideal de antivenenos modernos contra la picadura de alacrán. Revista Digital Universitaria. Volumen 15, número 11, 1º de noviembre de 2014. Patentes: Lidia Riaño, Everardo Rodriguez, Baltazar Becerril, Lourival Possani. Familia de variantes de anticuerpos recombinantes humanos que neutralizan a las toxinas Cn2 y Css2 de alacrán así como a los venenos respectivos: Centruroides noxius y Centruroides suffusus suffusus. Mx 2011/a/009885. Fecha de solicitud 21 de septiembre de 2011. Título de Patente No. 325627. Concedida el 13 de noviembre de 2014. A raíz de la divulgación de la concesión de esta patente, se dieron múltiples entrevistas a los medios locales de comunicación en radio, televisión y periódicos. También se concedieron entrevistas a la AMC y al Consejo Consultivo de Ciencias. Lidia Riaño Umbarila, Everardo Remi Rodríguez Rodríguez, Ilse Viridiana Gómez Ramírez, Jonathan Noé Arredondo López, Timoteo Celso Olamendi Portugal, Guillermo Fernández Taboada, Lourival Domingos Possani, Baltazar Becerril Luján. FAMILIA DE VARIANTES DE ANTlCUERPOS RECOMBINANTES HUMANOS QUE NEUTRALIZAN A LOS PRINCIPALES VENENOS DE ALACRANES MEXICANOS. Solicitada en el IMPI el 2 de septiembre de 2015. Títulos y autores de los carteles que presentará el grupo: 1. Generación de un scFv neutralizante de la toxina Css2 del alacrán Centruroides suffusus a partir del scFv C1. Everardo Remi Rodríguez, Lidia Riaño, Jonathan Arredondo, Guillermo Fernández, Timoteo Olamendi, Lourival Possani, Baltazar Becerril. 2. Generación de scFvs capaces de reconocer a la toxina Cll2 del alacrán Centruroides limpidus limpidus. Ilse Gómez, Lidia Riaño, Guillermo Fernández, Timoteo Olamendi, Lourival Possani, Baltazar Becerril. Grupo de Carlos F. Arias (2014-2015) Título: Virus emergentes. Resumen: En el periodo 2014-2015 mi laboratorio ha continuado profundizando en el mecanismo de los rotavirus para ingresar a célula polarizadas y no polarizadas, caracterizando en particular el papel de la ocludina (proteína que forma parte de las uniones estrechas intercelulares) en este proceso. Hemos también avanzado en la evaluación del papel del transporte vesicular entre el retículo endoplásmico y el aparato de Golgi en la morfogénesis rotavirus, así como la función del citoesqueleto de actina en el egreso del virus de la célula. Por otra parte, hemos caracterizado factores celulares necesarios para la eficiente replicación de astrovirus y hemos iniciado con la caracterización de la diversidad de receptores que pueden existir para diferentes serotipos de este virus, que al igual que los rotavirus, son causa importante de las gastroenteritis infantiles y se han asociado recientemente con problemas neurológicos. En estos dos años hemos también dedicado un esfuerzo importante a estudios metagenómicos para determinar: a) los patógenos virales asociados a enfermedades respiratorias moderadas y severas; b) la diversidad viral presente en las pozas del Valle de Cuatrociénegas y; c) la diversidad de retrovirus endógenos en murciélagos hematófagos y la evolución de receptores Toll-like en murciélagos, en colaboración con Marina Escalera, ex-estudiante de maestría del laboratorio. Dicho lo anterior, en esta ocasión mi presentación versará alrededor de virus emergentes. Las emergencias sanitarias causadas en 2009-2010 por la pandemia del virus de influenza H1N1 y en 2014-2015 por la severa epidemia de Ébola, que está en proceso en África Occidental, ha demandado una parte no menor de tiempo en la participación de comités de expertos universitarios, estatales y federales y en entrevistas a los medios de comunicación. Por esta razón y por la reciente llegada del virus de chikungunya a nuestro país y el inminente arribo del virus de Zika haré, no un reporte de mis actividades puntuales, sino una revisión general sobre algunos conceptos relacionados con la emergencia de nuevas enfermedades virales y sobre algunas acciones que estamos emprendiendo para contribuir a contender con estos problemas, que seguirán presentándose en el futuro. Integrantes del Grupo: Líderes Académicos Carlos F. Arias, Susana López Investigadores asociados Pavel Isa, Tomás López, Blanca Taboada, Carlos Sandoval Técnicos académicos Rafaela Espinosa, Marco A. Espinoza Estudiantes: Licenciatura: Fredy Peralta, Luis Eduardo Hernández Maestría: Cinthia Martínez, Yerli Marín, Ana Sánchez, Oscar Trejo, Nayeli Aguilar, Giovanni Elizarraras, Juan Manuel Carreño, Alma Valencia. Doctorado: Daniela Silva, Marco Aurelio Díaz, Jesús Torres, Andrea Murillo, Miguel A. Martínez, Rosa Ma. Rubio, Alfonso Oceguera, Liliana Sánchez, Luis Casorla, Joaquín Moreno, José Luis Martínez. Administrativos: Lorena Salazar, Miguel Angel Olvera. Nayeli Uribe Publicaciones: 1. Paulin LF, Soto-Del Río MD, Sánchez I, Hernández J, Gutiérrez-Ríos RM, López-Martínez I, Wong-Chew RM, Parissi-Crivelli A, Isa P, López S, Arias CF. 2014. PhyloFlu, a DNA microarray to determine the phylogenetic origin of influenza A gene segments and the genomic fingerprint of viral strains. J Clin Microbiol. 52:803-813. 2. Méndez E, Muñoz-Yañez C, Sánchez-San Martín C, Aguirre-Crespo G, Baños-Lara MD, Gutierrez M, Espinosa R, Acevedo Y, Arias CF, López S. 2014. Characterization of human astrovirus cell entry. J Virol. 88:2452-2460. 3. Díaz-Salinas MA, Silva-Ayala A, López S, and Arias CF. 2014. Rotaviruses reach late endosomes and require the cation-dependent mannose-6- phosphate receptor and the activity of cathepsin proteases to enter the cell. J Virol. 88:4389-4402. 4. Carrera Aguirre VM, Mercado García MC, Trujillo Ortega ME, Mendoza Elvira SE, Isa Haspra P, Paulin Paz LP, Arias Ortiz CF, Sánchez-Betancourta JI. 2014. Genetic changes detected in the internal genes of porcine influenza viruses isolated in Mexico. Veterinaria México Open Access 1(1) July-September, 1-22. 5. Escalera-Zamudio M, Nelson MI, Cobián-Güemes AG, López-Martínez I, Cruz-Ortiz N, Iguala-Vidales M, Rodríguez-García E, Barrera-Badillo G, Díaz-Quiñonez JA, López S, Arias CF, Isa P, and members of Colegio de Pediatría del Estado de Veracruz. 2014. Molecular epidemiology of influenza A/H3N2 viruses circulating in Mexico from 2003 to 2012. PLoS One. 30;9(7):e102453. doi: 10.1371 6. Taboada B, Espinoza MA, Aponte FE, Isa P, Arias-Ortiz MA, Monge-Martínez J, RodríguezVázquez R, Díaz-Hernández F, Zárate-Vidal F, Wong-Chew RM, Firo-Reyes V, del RíoAlmendárez CN, Gaitán-Meza J, Villaseñor-Sierra A, Martínez-Aguilar G, Salas-Mier MC, Noyola DE, Pérez-Gónzalez LF, López S, Santos-Preciado JI, Arias CF. 2014. Is there still room for novel viral pathogens in pediatric respiratory tract infections? PLoS One. 9(11):e113570. doi: 10.1371 7. Naccache SN, Federman S, Veeraraghavan N, Zaharia M, Lee D, Samayoa E, Bouquet J, Greninger AL, Luk KC, Enge B, Wadford DA, Messenger SL, Genrich GL, Pellegrino K, Grard G, Leroy E, Schneider BS, Fair JN, Martínez MA, Isa P, Crump JA, DeRisi JL, Sittler T, Hackett J Jr, Miller S, Chiu CY. 2014. A cloud-compatible bioinformatics pipeline for ultra-rapid pathogen identification from next-generation sequencing of clinical samples. Genome Research, 24 (7): 1180-92. Doi:10.1101/gr.171934 8. Arias CF, Silva-Ayala D, López S. 2015. Rotavirus entry, a deep journey into the cell with several exits. J Virol. 89(2):890-893. 9. Torres-Flores JM, Silva-Ayala D, Espinoza MA, López S, Arias CF. 2015. The tight junction protein JAM-A functions as coreceptor for rotavirus entry into MA104 cells. Virology, 475: 172178 10. Martínez MA, Soto-del Río MD, Gutiérrez RM, Chiu CY, Greninger AL, Contreras JF, López S, Arias, CF, Isa P. 2015. Parallel detection of gastrointestinal viruses by DNA microarray. J. Clin. Microbiol. 53:136-45. 11. Aponte FE, Taboada B, Espinoza MA, Arias-Ortiz MA, Monge-Martínez J, RodríguezVázquez R, Díaz-Hernández F, Zárate-Vidal F, Wong-Chew RM, Firo-Reyes V, del RíoAlmendárez CN, Gaitán-Meza J, Villaseñor-Sierra A, Martínez-Aguilar GM, García-Borjas M, Noyola DE, Pérez-Gónzalez LF, López S, Santos-Preciado JI, Arias CF. 2015. Rhinovirus is an important pathogen in upper and lower respiratory tract infections in Mexican children. Virology Journal. 12:31. DOI: 10.1186/s12985-015-0262-z. 12. Escalera-Zamudio M, Zepeda LM, Heeger F, Loza-Rubio E, Rojas-Anaya E, Méndez-Ojeda ML, Taboada B, Mazzoni CJ, Gilbert TM, Arias CF, and Greenwoodd AD. 2015. A novel endogenous betaretroviruses in the common vampire bat (Desmodus rotundus) suggests multiple independent infection and cross-species transmission events. J. Virol. 89:5180-4. 13. Wong-Chew RM, Espinoza MA, Taboada B, Aponte FE, Arias-Ortiz MA, Monge-Martínez J, Rodríguez-Vázquez R, Díaz-Hernández F, Zárate-Vidal F, Santos-Preciado JI, López S, Arias CF. 2105. Value of respiratory virus detection in symptomatic children in private physician office settings in five communities in the state of Veracruz, Mexico. BMC Res Notes. 25; 8(1):261. doi: 10.1186/s13104-015-1239-0 14. Murillo M, Vera-Estrella R, Barkla BJ, Méndez E, and Arias CF. 2015. Identification of host cell factors associated with astrovirus replication in Caco-2 cells. J. Virol. 89:10359-70. 15. López T, López S, Arias, CF. 2015. The tyrosine kinase inhibitor genistein induces the detachment of rotavirus particles from the cell surface. Virus Res. 210:141-148. 16. Torres-Flores JM, Arias CF.2015. Tight Junctions Go Viral! Viruses. 7(9):5145-54. doi: 10.3390/v7092865. 17. Sánchez-Tacuba L, Rojas M, Arias CF, López S. 2015. Rotavirus controls the activation of the OAS/RNase L pathway using at least two distinct mechanisms. J Virol pii: JVI.01874-15 18. Escalera-Zamudio M, Zepeda-Mendoza LM, Loza-Rubio E, Rojas-Anaya E, Méndez-Ojeda ML, Arias CF, Greenwood AD. 2015. The evolution of bat nucleic acid sensing Toll-like receptors. Mol Ecol. 2015 Oct 26. doi: 10.1111/mec.13431 Capítulos de libro: 19. Arias CF, López, T, Murillo A. 2015. Astrovirus replication and reverse genetics. In Viral Gastroenteritis: Molecular biology, pathogenesis, epidemiology and vaccine development Editors: Lennart Svensson, Ulrich Desselberger, Harry B Greenberg, and Mary K Estes. Elsevier Academic Press. En Prensa. 20. Arias CF, Silvia-Ayala D, Isa P, Díaz-Salinas MA, and López S. 2015. Rotavirus attachment, internalization, and vesicular traffic. In Viral Gastroenteritis: Molecular biology, pathogenesis, epidemiology and vaccine development Editors: Lennart Svensson, Ulrich Desselberger, Harry B Greenberg, and Mary K Estes. Elsevier Academic Press. En Prensa. 21. Arias CF, López T. 2015. Astrovirus. In Clinical Virology 4th Edition. Editors: Richman DD, Whitley RJ, Hayden FG. ASM Press. Washington DC. En Prensa Patentes: 1. Solicitud provisional USA: “A novel polymavirus associated with diarrhea in children”. Charles Chiu, Guixia Yu, Alexander Greninger, Pavel Isa, Carlos F. Arias, Joseph De Risi, Juliet Parsonnet y Steve Miller. No. 61/469,380. Licenciada a Abbot Laboratories. Alumnos graduados: Maestría 1. Fernando Aponte Sánchez. 2014. Caracterización de la diversidad genética de rinovirus circulantes en la población de México. Ciencias Bioquímicas/IBt, UNAM. (Tutor: Carlos Arias) 2. Juan Manuel Carreño. 2014. Caracterización de la infección de rotavirus en células MA104 pre-estimuladas con IFN tipo I y evaluación de la permisividad de distintas líneas celulares a la infección. Ciencias Bioquímicas/IBt, UNAM. (Tutor: Susana López) 3. Cinthia Martinez Sanchez. 2014. Caracterizacion de los gangliosidos involucrados en la interaccion de celulas MA104 con diferentes cepas de rotavirus. Ciencias Bioquímicas/IBt, UNAM. (Tutor: Pavel Isa) 4. José Luis Martínez Guevara. 2015. Análisis funcional de complejo proteico COPI en la infección por rotavirus. Ciencias Bioquímicas/IBt, UNAM. (Tutor: Carlos Arias) 5. Yerli Marin Tovar. 2015. Caracterización del procesos de traducción durante la infección por rotavirus. Ciencias Bioquímicas/IBt, UNAM. (Tutor: Susana López) Doctorado 6. Daniela Silva Ayala. 2014 (Junio 12). Identificación de genes celulares relevantes para la replicación de rotavirus. Ciencias Bioquímicas/IBt, UNAM. (Tutor: Carlos Arias) 7. Marco Aurelio Díaz Salinas. 2014 (Octubre 29). Estudio de los factores virales involucraden los mecanismos de endocitosis asociados a la entrada de rotavirus. Ciencias Bioquímicas/IBt, UNAM. (Tutor: Carlos Arias) 8. Miguel Angel Martinez Mercado. 2015. Desarrollo y validacion de un microarreglo de DNA para deteccion y subtipificacion de agentes virales asociados a gastroenteritis. Ciencias Bioquímicas/IBt, UNAM. (Tutor: Pavel Isa) Docencia: Carlos Arias 1. Curso “Virología Molecular”. Curso anual para maestría en el posgrado en Ciencias Bioquimicas- IBt/UNAM y la licenciatura en Biología de la Facultad de Ciencias, UNAM. Ago 2014 – Ene 2013. Y, Ago 2015 – Ene 2016. Responsables Carlos Arias y Tomás López. En este curso participan, además de los responsables, los siguientes investigadores del grupo: Susana López, Pavel Isa, Carlos Sandoval 2. Licenciatura en Ciencias Genómicas. Participación en curso: "Aplicaciones de la Genómica". 2014. Tema: Metagenómica de enfemedades respiratorias (3h). 3. Licenciatura en Ciencias Genómicas. Participación en curso: "Fronteras en Genómica". 2014. Tema: Ébola (3h). 4. Licenciatura en Ciencias Genómicas. Curso: Técnicas Experimentales 2015 (120 h). Susana López 5. Licenciatura en Ciencias Genómicas. Curso: Técnicas Experimentales 2015 (120 h). Pavel Isa 6. Curso Básico “Biología celular” en Programa en Ciencias Bioquímicas de la UNAM. Tema: Matriz extracelular. 2014 7. Participación en 4tas Jornadas Académicas de Químicos del INER. Nivel posgrado. Impartido en Instituto Nacional de Enfermedades Respiratorias. 2014 8. Licenciatura en Ciencias Genómicas. Participación en curso: "Aplicaciones de la Genómica". 2015. Tema: Virología y Genómica (3h). Blanca Taboada 9. Bases de programación. Lenguaje Perl 2015-2, Programas de Doctorado en Ciencias Bioquímicas (PDCBioq) y Biomédicas (PDCBiomed), UNAM. Se impartió en el CCG. Donativos: 1. Proyecto DGAPA/UNAM. IN211411-3 (2011-2014). Mecanismos de evasión de la respuesta antiviral por rotavirus. Responsable: Susana López. 2. Proyecto. SEP-CONACYT 153639 (2011-2016). Caracterización de las interacciones de los rotavirus con su célula huésped necesarias para el establecimiento de una infección productiva. Responsable: Susana López. 3. Proyecto DGAPA/UNAM. IN207313 (2013-2015). Identificación de los gangliósidos específicos involucrados en la entrada de diversas cepas de rotavirus. Responsable: Pavel Isa. 4. Proyecto DGAPA/UNAM. IG200114 (2014-2016). Biología celular de la infección por rotavirus. Responsable: Carlos Arias. 5. Proyecto. SEP-CONACYT CB-221019 (2014-2016). Caracterización del tráfico vesicular de los rotavirus durante su replicación en la célula hospedera. Responsable: Carlos Arias. Premios y distinciones: Premio Nacional de Ciencias y Artes 2014 en el área de Ciencias Físico-Matemáticas y Naturales. Títulos y autores de los carteles que presentará el grupo: 1. Pharmacological inhibitors of COPI/Arf1 prevent assembly of the outer layer protein of rotavirus and trimerization of the surface glycoprotein VP7. José Luis Martínez, Catherine Eichwald, Francesca Arnoldi, Oscar Burrone, Susana López, Carlos F. Arias. 2. Rotavirus entry and release is polarized in a small intestine cell line. Pavel Isa, Diego Cevallos, Susana Lopez, Carlos F. Arias
© Copyright 2026