GAUSSIAN 94 Archivo input de Gaussian 94
Curso de Modelado Molecular I
GAUSSIAN 94
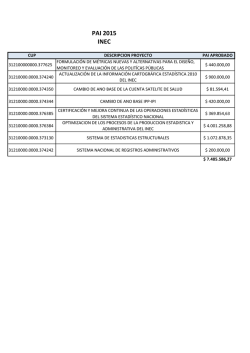
Este es un programa típico y sumamente extendido para realizar cálculos de estructura
electrónica. Aún cuando es posible realizar cálculos MM y semiempíricos, su empleo más
frecuente es en el cálculo ab initio Hartree-Fock, post-Hartree-Fock y DFT.
Gaussian, es un sistema de programas conectados para ejecutar una variedad de cálculos de
estructura electrónica. Es capaz de predecir muchas propiedades moleculares y reacciones
químicas, incluyendo:
• Energías y estructuras moleculares
• Energías y estructuras de estados de transición
• Frecuencias vibracionales
• Espectros IR y Raman
• Propiedades termodinámicas
• Energías de reacción y de enlace
• Caminos de reacción
• Orbitales moleculares
• Cargas atómicas
• Momentos multipolares
• Apantallamiento en Resonancia Magnética Nuclear y susceptibilidad magnética
• Afinidades electrónicas y potenciales de ionización
• Polarizabilidad e hiperpolarizabilidad
• Potenciales electrostáticos y densidades electrónicas
Los cálculos pueden realizarse para sistemas en fase gaseosa y en solución, en sus estados
fundamentales y excitados. Así, Gaussian permite explorar áreas de interés químico como efectos
del sustituyente, mecanismos de reacción, superficies de energía potencial, energías de excitación,
etc.
A diferencia de Hyperchem, Gaussian es un programa que no trabaja con un entorno gráfico.
Consecuentemente, la información sobre que tipo de trabajo efectuar y como realizarlo debe
entrarse manualmente, lo cual, en principio, requiere más atención de parte del usuario que en un
trabajo del tipo Hyperchem.
Archivo input de Gaussian 94
El input de Gaussian 94 (g94 de ahora en adelante) consiste en una serie de líneas de un
archivo de texto ASCII. La estructura básica de un archivo input de g94 incluye diferentes
secciones:
Sección del archivo input
Comandos Link 0
Ruta de cálculo (líneas #)
Sección de título o comentarios
Especificación de la estructura molecular
Propósito
Localiza y asigna un nombre a los archivos checkpoint
(entre otras posibles funciones)
Especifica el tipo deseado de cálculo, el modelo
químico y las opciones
Breve descripción del cálculo
Especifica el sistema molecular en estudio
1
Curso de Modelado Molecular I
A continuación se muestra un ejemplo de un archivo input, en el que se indica una
optimización de la geometría de la molécula de hidrógeno utilizando el método B3LYP
(funcionales de la densidad) con la base 3-21G, y, un cálculo posterior de frecuencias. Notar que
en el sistema operativo en que se realiza el cálculo, se debe iniciar el archivo input ejecutando el
comando para correr G94 ($RunGauss).
$RunGauss
%Chk=hidrogeno.chk
(línea en blanco)
#P B3LYP/3-21G OPT=z-mat Freq
(línea en blanco)
Optimización de la molécula de hidrógeno y cálculo de frecuencias
(línea en blanco)
01
H
H 1 dHH
(línea en blanco)
dHH=0.9
(al final del archivo se debe dejar dos líneas
en blanco)
Debe salvarse como un
archivo
del
tipo
nombre.dat
En este caso
llamarse h2.dat.
podría
COMANDOS LINK 0:
Estos comandos son opcionales y preceden a la ruta de cálculo en el caso en que estén
presentes.
%Chk=nombre.chk
Localiza y asigna un nombre a los archivos checkpoint.
Estos archivos resultan de gran utilidad en aquellos
casos en donde se debe reiniciar un cálculo.
RUTA DE CÁLCULO:
La ruta es el conjunto de comandos que definen el cálculo y que permite especificar
completamente la forma en que el mismo se efectuará. Especifica el tipo de trabajo que se quiere
correr, el método teórico, el set de bases que se utilice, etc. Todos estos aspectos de especifican
por palabras clave.
La sección de ruta empieza obligatoriamente con un # y es terminada con una línea en
blanco. Pueden darse varias líneas de palabras clave. En la mayoría de los casos no importa en que
orden se den las palabras clave, excepto que estén relacionadas específicamente en lo que sigue.
2
Curso de Modelado Molecular I
Formas alternativas
#
Resultado
Output normal
#P
Se genera un output adicional. Incluye mensajes al comienzo
y al final de cada link, así como información de la
convergencia SCF.
#T
El output se reduce a la información adicional y a los
resultados (“terse output”)
Luego se debe especificar lo que se quiere hacer, si un cálculo simple, una optimización de
geometría, la búsqueda de un estado de transición, el cálculo de frecuencias de vibración, etc.
ESPECIFICACIÓN DEL CÁLCULO
TIPO DE CÁLCULO: Define el tipo de cálculo que se desea utilizar. Dependiendo del tipo de
cálculo que desee efectuarse, g94 tiene una serie de palabras clave que básicamente pueden
clasificarse en los siguientes grupos:
1. Cálculo de la energía y de la función de onda únicamente. Este es el cálculo que se
realiza en caso que no se estipule alguna otra palabra clave. Si bien no es necesario
identificarlo explícitamente, el tipo de cálculo se reconoce por la palabra clave SP.
2. Cálculos de superficies de energía potencial. Existen dos tipos básicos de cálculo,
aquéllos en los que se recorre algún camino de reacción y que se reconocen con la
palabra clave IRC y aquéllos en los que se calcula una superficie de energía
potencial reducida o global y que se identifican con la palabra clave SCAN. Tanto
aquí como en el apartado anterior es crucial considerar cuál va a ser el modelo
computacional que se empleará para generar la energía y la función de onda.
3. Cálculos de optimización de geometría. Básicamente, estos cálculos se reconocen
con la palabra clave OPT. Es posible determinar únicamente la fuerza sobre los
núcleos, sin proceder a realizar una optimización de geometría, y para ello se
emplea la palabra clave FORCE. Si quiere realizarse efectivamente la
optimización, entonces es posible optimizar hacia un mínimo, con lo cual OPT no
necesita ninguna cualificación especial, o hacia un punto de ensilladura de primer
orden (aproximación a un estado de transición) para lo que se usa la combinación
de keywords OPT=TS. Si se desea realizar una generación automática del eventual
estado de transición y luego proceder a la optimización, se usa la combinación
OPT=QST2. Finalmente, para chequear el tipo de punto estacionario o crítico que
se ha encontrado, se calculan las derivadas segundas de la energía, para lo que se
usa la palabra clave FREQ en un cálculo por separado sobre la geometría
optimizada, o la combinación OPT FREQ para que proceda al cálculo de la
frecuencia posteriormente a la optimización de geometría. En este apartado, al igual
que los anteriores, necesitamos conocer cual es el modelo computacional a
emplear, pero también existen ciertas consideraciones especiales acerca de la
metodología de optimización de geometría.
3
Curso de Modelado Molecular I
4. Cálculo de propiedades. Si bien algunas propiedades (cargas, momentos dipolares,
etc.) se calculan por defecto, existen algunas otras que deben implicarse
específicamente y por lo tanto definen tipos de cálculo. En otra sección de este
texto se mostrará cuáles son las palabras claves necesarias para el cálculo de las
propiedades electrónicas más frecuentes.
MODELOS COMPUTACIONALES:
Los modelos computacionales se definen por el método de cálculo que se emplea y las
características particulares del mismo (por ejemplo, el conjunto de base). En general, un
modelo computacional se va a identificar como:
MÉTODO(modificadores)/{conjunto de base}
No necesariamente (y de ahí las llaves) {conjunto de base} debe estar presente. En lo que
sigue se va a describir los distintos tipos de cálculo posibles en g94 y cuales son las palabras
clave que los identifican. Antes de hacerlo, sin embargo, se verá cuales son las posibilidades
de conjuntos de funciones de base, pues ya se mencionó que fundamentalmente se va a
emplear g94 para cálculos ab initio y DFT.
Conjuntos de funciones de base
Los conjuntos de funciones de base disponibles en g94 se identifican en la siguiente tabla
Palabra clave
Significado
STO-3G
Base mínima STO-3G
3-21G, 4-31G, 6-21G, 6-31G
Bases split-valence p-q1G
D95, D95V
Bases DZ de Dunning-Huzinaga
respectivamente)
(aug-)cc-pVxZ
Bases correlation-consistent con x=D,T,Q,5
6-311G
Base 6-311G para los átomos de la primera fila y
modificada para la 3ra fila, McLean-Chandler para la 2da,
Wachters-Hay para la 1ra de transición
+,++
Agrega funciones difusas sólo a los átomos principales o
también a los hidrógenos
*,**
Agrega un conjunto de funciones de polarización a los
átomos principales o también a los hidrógenos
(x,y)
Agrega x funciones de polarización en los átomos
principales, y en los hidrógenos (p.ej., (3df,3pd)).
GEN
Lee del archivo de entrada el conjunto de base que se
proporciona en forma explícita (Gen=general).
(full
y
valence
4
Curso de Modelado Molecular I
A continuación del modelo computacional pueden agregarse ciertas palabras claves que
actúan sobre el conjunto de funciones de base. Las mas frecuentemente usadas son:
ExtraBasis: Agrega bases extras (que se leen del input) al conjunto de base standard
escogido.
GFInput: Imprime el conjunto de funciones de base en forma adecuada para ser
entrado como base general.
5D, 7F: implica que se usan funciones de polarización en forma polar (i.e., eliminando
las funciones linealmente dependientes).
6D, 10F: implica que se usan gaussianas cartesianas. F simboliza también a las
funciones de orden superior.
MÉTODOS DE CÁLCULO:
Por una parte tenemos los métodos de cálculo que no requieren la especificación de un
conjunto de base:
•
Métodos Semiempíricos: el g94 puede utilizar las palabras claves CNDO, INDO,
MINDO3, MNDO, AM1, PM3 y PM3MM para identificar los respectivos
hamiltonianos semiempíricos (PM3MM identifica al método PM3 con una
corrección obtenida mediante Mecánica Molecular).
Por otra parte, tenemos los métodos ab initio que sí requieren la especificación de un
conjunto de funciones de base. Estos métodos pueden ser de tres tipos:
•
Métodos Hartree-Fock: En este caso tenemos pocas posibilidades
•
HF - Empleado para realizar cálculos Hartree-Fock simples, puede ser
invocado como restricto (RHF para configuraciones de capa cerrada y
ROHF para configuraciones de capa abierta) o irrestricto (UHF para los dos
casos).
•
CASSCF(n,m) - Empleado para realizar cálculos SCF de espacio activo
completo, se utiliza cuando el estado a investigar no puede ser representado
por un único determinante de Slater. El CASSCF admite muchos
modificadores, pero los dos mas importantes son (n,m) que indica usar n
electrones en m orbitales para el espacio activo, y NROOT=k que permite
calcular la energía para la k-ésima raíz (estado excitado).
•
GVB(n) - Se emplea similarmente a CASSCF, sólo que debido a la
estructura de esta función de onda, únicamente hay que proporcionarle el
número de orbitales n que estarán acoplados ("perfect-pairing" en la jerga
particular de GVB). Un caso particular de GVB es OSS que permite realizar
un cálculo de singulete de capa abierta (i.e. exactamente dos electrones con
acoplamiento singulete en dos orbitales ortogonales).
5
Curso de Modelado Molecular I
•
•
Métodos post-Hartree-Fock: En este caso, tenemos diversas posibilidades, de
acuerdo a que incluyamos la energía de correlación con métodos de CI,
perturbativos o de clusters acoplados.
•
CID o CISD - Teoría de interacción de configuraciones (CI) incluyendo
sólo dobles excitaciones (D), o también simples (SD). En estos casos, como
en los que siguen, incluir luego de la keyword que identifica al método, FC
entre paréntesis, indica que los electrones de core no se correlacionan,
mientras que incluír Full entre paréntesis, indica que todos los electrones
son correlacionados.
•
QCISD - la versión "size-extensive" de la CI, incluye sólo simples y dobles
excitaciones. Para incluir las excitaciones triples o triples y cuádruples en
forma no iterativa, se emplea QCISD(T) o QCISD(TQ) respectivamente. En
este último caso, se pueden incluir las triples en la evaluación de la MP4,
mediante QCISD(TQ,E4T).
•
MPn - métodos perturbativos de Møller-Plesset, de orden n (= 2, 3, 4 ó 5).
En este caso es importante señalar que MPn=Direct fuerza a la ejecución de
un algoritmo directo para ahorrar espacio en disco (a costa del tiempo de
cálculo para moléculas pequeñas pero muy efectivo para moléculas
grandes).
•
CCD o CCSD - Métodos de clusters acoplados, incluyendo doble
excitaciones o simples y dobles.
Métodos de funcionales de la densidad. En estos casos el método se forma por
una keyword que identifica al potencial de intercambio y otra que indica el
potencial de correlación. Los métodos mas frecuentemente usados se identifican
por las palabras claves LSDA (local-spin-density-approximation, equivalente a
SVWN) es un método sin gradientes, BP86, BPW91, BLYP identifican métodos de
gradiente, con el potencial de intercambio de Becke (1988) y los potenciales de
correlación de Perdew (1981), Perdew-Wang (1991) y Lee-Yang-Parr (1988).
Finalmente, B3LYP y B3PW91 identifican a los métodos de gradiente que incluyen
parte del intercambio exacto de Hartree-Fock (métodos ACM, adiabaticallycoupled-functionals).
Y, finalmente, tenemos los métodos especiales que permiten estudiar estados excitados
(CIS, CASSCF MP2, CIS MP2) o calcular afinidades electrónicas y potenciales de
ionización (OVGF), e, incluso, calcular el efecto del disolvente (SCRF=PCM).
Las distintas propiedades moleculares pueden obtenerse directamente en un output normal o
pueden necesitar palabras claves especiales. A continuación se como se estudian distintas
propiedades de importancia.
• Cargas atómicas - el análisis de Mulliken, que proporciona las cargas puntuales usadas
normalmente, se obtiene simplemente con POP (análisis poblacional). El análisis
poblacional puede realizarse de distintas formas que se describen a continuación
• POP=REG - análisis poblacional normal, imprime el análisis de Mulliken y los 5
orbitales ocupados más altos y desocupados mas bajos respectivamente
• POP=FULL - lo mismo, pero imprimiendo todos los orbitales
• POP=NONE - suprime completamente todo lo anterior
6
Curso de Modelado Molecular I
•
•
•
•
•
•
•
•
•
•
•
POP=CHARGE - ajusta las cargas al potencial electrostático para cada centro
atómico
• POP=(CHARGE,ATOMDIPOLE) - lo mismo que lo anterior pero ajusta también
los dipolos
• POP=MK - ajusta las cargas usando el esquema Merz-Kollman/Singh
• POP=NBO - realiza un estudio de orbitales naturales de enlace (mejores cargas
que Mulliken)
• POP=NATURALORB - realiza una descomposición en orbitales naturales de la
densidad total
• POP=NATURALSPIN - lo mismo pero para cada espín por separado
Multipolos - el momento dipolar y multipolos de mayor orden se obtienen simplemente
del análisis poblacional (POP)
Teoría de Bader - empleando la palabra clave AIM es posible realizar un análisis de
Bader de la densidad electrónica
Afinidades electrónicas y potenciales de ionización - Se obtienen usando la palabra
clave OVGF
PROP - permite obtener propiedades electrostáticas
NMR - permite obtener el espectro NMR
FREQ - permite obtener los espectros IR y Raman
CIS - permite obtener el espectro UV-Vis
POLAR - permite obtener las polarizabilidades e hiperpolarizabilidades
CUBE=DENSITY - permite obtener la densidad electrónica en una grilla de puntos
CUBE=POTENTIAL - permite obtener el potencial electrostático en una grilla de puntos
SECCIÓN DE TÍTULOS O COMENTARIOS:
La sección de títulos y comentarios consiste en una o más líneas de información descrptiva
del trabajo. Esta sección finaliza con una línea en blanco.
ESPECIFICACIÓN DE LA ESTRUCTURA MOLECULAR:
Comienza con la especificación de la carga y la multiplicidad de espin, que deben
indicarse como números enteros en la primera línea de esta sección. La carga de una molécula es
un número entero positivo o negativo, que especifica la carga total en una molécula. Así, 1 se usa
para un catión monocargado, -1 designa a un anión monocargado y 0 representa a una molécula
neutra. La multipicidad de espin para una molécula esta dada por la ecuación 2S +1, siendo S el
espin total de la molécula. Los electrones pueden tener espin alfa (+1/2) o beta (-1/2). Los
electrones apareados son un par alfa-beta, por lo que no contribuyen a la multiplicidad de espin.
Cada electrón desapareado contribuye en +1/2 a S (el espin total). Así, para un sistema que no
tiene electrones desapareados, la multiplicidad de espin es uno, y es un singulete. Un doblete (un
electrón desapareado) tiene multiplicidad de espin 2. Un triplete (dos electrones desapareados del
mismo espin) tiene multiplicidad de espin 3, y así sucesivamente.
La especificación de la estructura molecular puede ser realizada de varias formas.
Construyendo la matriz Z a mano, ingresando coordenadas cartesianas obtenidas de la literatura o
de resultados de un cálculo previo, de coordenadas generadas por o convertidas de un programa
gráfico (como Hyperchem), a partir de un archivo PDB, etc.
7
Curso de Modelado Molecular I
Una vez escrito el archivo input (en otra sección se verá una guía de uso del editor vi),
debe salvarse. En este caso se salvará como h2.dat. Luego deberá correrse el cálculo g94 de la
siguiente forma:
Makejob g94 h2.dat
g94 calcula lo que se indica en el archivo input y genera un archivo output con el nombre
h2.log, que contiene la información generada por el cálculo computacional (si el input se llama
nombre.dat, se genera un output nombre.log).
BREVE DESCRIPCIÓN DEL ARCHIVO OUTPUT
Se describirán algunas secciones del archivo que contiene la salida (el resultado) del
cálculo solicitado a g94.
----------------------------#P B3LYP/3-21G Opt=Z-Mat Freq
----------------------------------------------------------------------optimizacion de h2 y calculo de frecuencias
------------------------------------------Symbolic Z-matrix:
Charge = 0 Multiplicity = 1
H
H
1
dHH
Variables:
dHH
0.9
En esta parte, se muestra la ruta de cálculo, la sección de títulos o comentarios y la
especificación de la geometría molecular que contiene el archivo input.
Luego de un output inicial, g94 despliega una sección como la que se muestra a
continuación, para cada paso (step) de búsqueda de un mínimo en la superficie de energía
potencial (optimización de geometría).
8
Curso de Modelado Molecular I
GradGradGradGradGradGradGradGradGradGradGradGradGradGradGradGradGradGrad
Berny optimization.
Search for a local minimum.
Step number
1 out of a maximum of 20
All quantities printed in internal units (Hartrees-Bohrs-Radians)
Second derivative matrix not updated -- first step.
The second derivative matrix:
dHH
dHH
0.23575
Eigenvalues --0.23575
RFO step: Lambda=-1.91420721E-02.
Linear search not attempted -- first point.
Variable
Old X
-DE/DX
Delta X
Delta X
Delta X
New X
(Linear)
(Quad)
(Total)
dHH
1.70075 -0.06985
0.00000 -0.27404 -0.27404
1.42671
Item
Value
Threshold Converged?
Maximum Force
0.069851
0.000450
NO
La primera
línea (GradGradGrad)
es una línea
de separación
RMS
Force
0.069851
0.000300
NO entre diferentes pasos.
Maximum Displacement
0.274040
0.001800
NO
RMS
Displacement
0.274040
0.001200
NO
Predicted change in Energy=-8.852269E-03
GradGradGradGradGradGradGradGradGradGradGradGradGradGradGradGradGradGrad
Luego se muestra el algoritmo de optimización (Berny). Se indica que se realiza la
búsqueda de un mínimo local (Search for a local minimum) y el número de pasos
realizados. (Step number 1 out of a maximum of 20).
En la línea que comienza con la palabra Variable, indica los valores anteriores (Old) y
los nuevos (New) para las variables estructurales (distancias, ángulos y ángulos de torsión) en
unidades atómicas (bohrs y radianes).
A continuación se indica el resultado del test de convergencia. Cuando los cuatro valores
en la columna Converged? dicen Yes, entonces la optimización está completa y ha convergido,
presumiblemente a un mínimo local. Finalmente, se despliega el cambio en la energía, aunque este
no es un criterio de convergencia.
Item
Value
Threshold Converged?
Maximum Force
0.000167
0.000450
YES
RMS
Force
0.000167
0.000300
YES
Maximum Displacement
0.000438
0.001800
YES
RMS
Displacement
0.000438
0.001200
YES
Predicted change in Energy=-3.553019E-08
Optimization completed.
-- Stationary point found.
---------------------------!
Optimized Parameters
!
! (Angstroms and Degrees) !
------------------------------------------!
Name
Value
Derivative information (Atomic Units)
!
-----------------------------------------------------------------------!
dHH
0.746
-DE/DX =
0.0002
------------------------------------------------------------------------
Cuando la optimización converge, se muestra la estructura final (distancias, ángulos y ángulos de
torsión) en Angstroms y grados.
9
Curso de Modelado Molecular I
**********************************************************************
Population analysis using the SCF density.
**********************************************************************
Orbital Symmetries:
Occupied (SGG)
Virtual
(SGU) (SGG) (SGU)
The electronic state is 1-SGG.
Alpha occ. eigenvalues --0.42876
Alpha virt. eigenvalues -0.11703
0.71249
1.21970
Condensed to atoms (all electrons):
1
2
1 H
0.598711
0.401289
2 H
0.401289
0.598711
Total atomic charges:
1
1 H
0.000000
2 H
0.000000
Sum of Mulliken charges=
0.00000
Atomic charges with hydrogens summed into heavy atoms:
1
1 H
0.000000
2 H
0.000000
Sum of Mulliken charges=
0.00000
Electronic spatial extent (au): <R**2>=
5.0738
Charge=
0.0000 electrons
Dipole moment (Debye):
X=
0.0000
Y=
0.0000
Z=
0.0000 Tot=
0.0000
Quadrupole moment (Debye-Ang):
XX=
-1.9734
YY=
-1.9734
ZZ=
-1.5413
XY=
0.0000
XZ=
0.0000
YZ=
0.0000
Octapole moment (Debye-Ang**2):
XXX=
0.0000 YYY=
0.0000 ZZZ=
0.0000 XYY=
0.0000
XXY=
0.0000 XXZ=
0.0000 XZZ=
0.0000 YZZ=
0.0000
YYZ=
0.0000 XYZ=
0.0000
Hexadecapole moment (Debye-Ang**3):
XXXX=
-1.7209 YYYY=
-1.7209 ZZZZ=
-2.6632 XXXY=
0.0000
XXXZ=
0.0000 YYYX=
0.0000 YYYZ=
0.0000 ZZZX=
0.0000
ZZZY=
0.0000 XXYY=
-0.5736 XXZZ=
-0.7420 YYZZ=
-0.7420
XXYZ=
0.0000 YYXZ=
0.0000 ZZXY=
0.0000 -----------------------------------------------------------------------
Un análisis poblacional de Mulliken sigue a continuación de los resultados de energía SCF,
este análisis particiona la carga en la molécula por átomo (por defecto, g94 realiza un análisis
poblacional de Mulliken, si se desea calcular las cargas puntuales sobre los átomos mediante otros
métodos se debe especificar en la línea correspondiente a la ruta de cálculo). La sección que
comienza con Total atomic charges, indica la carga total estimada en cada átomo de la
molécula (cero para cada átomo de hidrógeno en este caso). Luego se indica el momento dipolar y
momentos mayores (cuadrupolares, etc.).
10
Curso de Modelado Molecular I
Harmonic frequencies (cm**-1), IR intensities (KM/Mole),
Raman scattering activities (A**4/AMU), Raman depolarization ratios,
reduced masses (AMU), force constants (mDyne/A) and normal coordinates:
1
SGG
Frequencies -- 4474.3810
Red. masses -1.0078
Frc consts -11.8878
IR Inten
-0.0000
Raman Activ -0.0000
Depolar
-0.0000
Atom AN
X
Y
Z
1
1
0.00
0.00
0.71
2
1
0.00
0.00 -0.71
g94 predice las frecuencias y las intensidades, lo cual permite simular los espectros IR de
las estructuras en estudio, así como caracterizar los puntos estacionarios encontrados.
Las optimizaciones de geometría convergen a una estructura en la superficie de energía
potencial donde las fuerzas en el sistema son esencialmente cero. La estructura final puede
corresponder a un mínimo en la superficie de energía potencial, o puede representar a un punto de
ensilladura, el cual es un mínimo con respecto a algunas direcciones en la superficie y un máximo
en una o más direcciones. Un punto de ensilladura de primer orden –el cual es un máximo en una
única dirección y un mínimo en todas las otras direcciones –corresponde a una estructura de
transición que conecta dos mínimos.
Frecuencias encontradas
0 frecuencias imaginarias
1 frecuencia imaginaria
Significado
A continuación se debe
Comparar la energía de esta
geometría
con
otras
La estructura corresponde a un
correspondientes a la misma
mínimo
estructura si se desea encontrar
el mínimo global
Determinar si esta estructura
conecta los reactivos y
La estructura corresponde a una productos examinando los
estructura de transición
modos
normales
de
la
frecuencia
imaginaria
o
realizando un cálculo IRC.
11
Curso de Modelado Molecular I
\\#P GEOM=AL
LCHECK GUESS=TCHECK RB3LYP/3-21G FREQ\\optimizacion de h2 y calculo de
frecuencias\\0,1\H\H,1,dHH\\dHH=0.74599171\\Version=Cray-C90-Unicos-G
98RevA.7\State=1-SGG\HF=-1.170611\RMSD=1.127e-13\RMSF=9.638e-05\Dipole
=0.,0.,0.\DipoleDeriv=0.,0.,0.,0.,0.,0.,0.,0.,0.,0.,0.,0.,0.,0.,0.,0.,
0.,0.\Polar=0.,0.,0.,0.,0.,6.1331249\PG=D*H [C*(H1.H1)]\NImag=0\\-0.00
012115,0.,-0.00012115,0.,0.,0.38177943,0.00012115,0.,0.,-0.00012115,0.
,0.00012115,0.,0.,-0.00012115,0.,0.,-0.38177943,0.,0.,0.38177943\\0.,0
.,0.00016694,0.,0.,-0.00016694\\\@
WE ARE REACHING THE STAGE WHERE THE PROBLEMS WE MUST SOLVE ARE
GOING TO BECOME INSOLUBLE WITHOUT COMPUTERS.
I DO NOT FEAR COMPUTERS. I FEAR THE LACK OF THEM.
-- ISSAC ASIMOV
Job cpu time: 0 days 0 hours 0 minutes 36.0 seconds.
File lengths (MBytes): RWF=
11 Int=
0 D2E=
0 Chk=
8 Scr=
1
Normal termination of Gaussian 98.
Al final del archivo output, se resumen los resultados. Entre ellos, puede verse la energía
de la estructura optimizada (HF=-1.170611). Un trabajo de Gaussian exitoso finaliza siempre
con una cita elegida al azar. El tiempo de CPU e información del uso de otros recursos se
presentan antes de finalizar el archivo.
Cabe aclarar que esta breve descripción del output incluye exclusivamente los datos
relevantes para el ejemplo que se analiza (optimización de la molécula de hidrógeno y el cálculo
de frecuencias).
12
© Copyright 2026